Compêndio de métodos e de boas práticas em coleção de cultura de leveduras do Instituto de Biologia do Exército
Compendium of methods and good practices in yeast culture collection of the Brazilian Army Biology Institute
Marcos Dornelas Ribeiro1
Eliane Olmo Pinheiro2
Alberto Magno Lobo Colares3
Andre Luis Meriano Figueiredo3
Vanusa Guimarães Dutra1
Caleb Guedes Miranda dos Santos4
Vanusa Guimarães Dutra5
1Doutor(a) em microbiologia Instituto de Biologia do Exército – Rio de Janeiro-RJ, Brasil.
2Bacharel em Farmácia Bioquímica/UERJ – Estudante de Mestrado. Brasil.
3Farmacêutico-Bioquímico – Instituto de Biologia do Exército – Rio de Janeiro-RJ, Brasil.
4Doutor em Ciências Biológicas – Biofísica – Instituto de Biologia do Exército – Rio de Janeiro-RJ, Brasil.
5Prof. Doutor em Vigilância Sanitária – Universidade Federal do Rio de Janeiro (UFRJ)/Univ. do Estado do Rio de Janeiro (UERJ). Rio de Janeiro-RJ, Brasil.
Instituição: Instituto de Biologia do Exército – Rio de Janeiro-RJ, Brasil.
Recebido em 28/02/2019
Artigo aprovado em 05/04/2019
DOI: 10.21877/2448-3877.201900828
INTRODUÇÃO
Ao final do século XIX, Ismael da Rocha e Oswaldo Cruz concretizaram o sonho de ver instalado no Brasil um laboratório de pesquisa aos moldes do Instituto Pasteur, da França. Em 19 de dezembro de 1894 foi assinado pelo Presidente Prudente de Moraes o decreto de nº 1.915 criando o Laboratório Militar de Microscopia Clínica e Bacteriologia aos moldes do Instituto francês. As atividades iniciaram em 2 de julho de 1896, na rua atualmente conhecida como Senador Furtado, no bairro do Maracanã, na cidade do Rio de Janeiro. Na transição do ano de 1898 para 1899, suas instalações foram transferidas para a Rua General Canabarro 40, também no bairro do Maracanã, permanecendo nesse local até 1907. A partir dessa data, o laboratório passou a funcionar no Pavilhão Rodrigues Alves do Hospital Central do Exército (HCE), na rua Francisco Manuel, no bairro de Triagem. Posteriormente teve nova nomenclatura: Laboratório Militar de Bacteriologia. Assim permaneceu até o período provisório do governo de Getúlio Vargas, que em 13 de janeiro de 1932, por meio do Decreto de nº 20.943, deu-lhe nova denominação: Instituto Militar de Biologia, momento no qual adquiriu, em definitivo, seu atual endereço no bairro de Triagem, no Rio de Janeiro. Por fim, em 12 de abril de 1943, recebeu a denominação Instituto de Biologia do Exército.
Hoje ocupa três regiões distintas: a sede, em Triagem, a sua área de produção veterinária, no Campo de Instrução de Gericinó (CIG), e parte do prédio anexo do Hospital Geral do Rio de Janeiro (HGeRJ) com o posto avançado de captação de sangue e coleta de sangue para exames laboratoriais.
A estrutura organizacional do IBEx é composta pelas Divisões Técnica, Veterinária, Banco de Sangue e Soros e Vacinas, Ensino e Pesquisa, Administrativa, além das assessorias de Gestão da Qualidade e de Vigilância em Saúde.
A Divisão de Ensino e Pesquisa (DEP) do IBEx, desde a origem do Instituto até os dias atuais, vem se sobressaindo no campo de formação técnica e investigação científica na área da Saúde. No ensino, contribui realizando cooperações de instrução com diversos cursos militares operacionais, além de abrigar três cursos do Programa de Capacitação de Militares de Saúde (Procap/SAU) da Diretoria de Saúde, promovendo a atualização dos militares desse Serviço. Na pesquisa, o resultado de seus experimentos e produção científica tem sido divulgado em publicações internacionais em áreas como Microbiologia, Defesa Biológica, Genética Humana e Genética Forense, possibilitando a criação do Mestrado em Defesa Biológica, reconhecido pela Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES). Somado a isso, o IBEx adequou e modernizou seus laboratórios de pesquisa, criando o Centro de Estudos em Biodefesa, que dispõe de um laboratório com nível de contenção biológica superior e que gerencia o laboratório de Biossegurança NB-3, componente importante do Sistema de Defesa Química, Biológica, Radiológica e Nuclear do Exército (SisDQBRNEx).
Tendo em vista a necessidade de organização para atender às demandas da defesa biológica, o IBEx vem buscando sua capacitação, adequação e estruturação para dominar a tecnologia de identificação e manejo dos microrganismos envolvidos em bioterrorismo, tanto no que tange às metodologias quanto no que se refere a equipamentos e estrutura física. Nesse contexto, o Exército Brasileiro tem feito aquisições de equipamentos e executado projetos como a construção de um Laboratório de Defesa Biológica (LDB), com nível 3 de Biossegurança (NB3), no IBEx, como mencionado anteriormente, para a caracterização de agentes envolvidos em guerra biológica e bioterrorismo. O LDB deverá servir de base técnico-científica em âmbito nacional para estudos e apoio a outras instituições (públicas e privadas) comprometidas direta ou indiretamente com a segurança nacional e desenvolvimento de pesquisas.
O IBEx mantém hoje estreito relacionamento com instituições renomadas de pesquisas no cenário nacional, a partir do convênio com a Fundação Oswaldo Cruz (Fiocruz), através do Laboratório de Fisiologia e Controle de Artrópodes e Vetores (Laficave), dos Programas de Incentivo à Pesquisa em Defesa Nacional (Pró-Defesa III) com projetos entre o IBEx e a Universidade Federal do Rio de Janeiro e a Universidade do Estado do Rio de Janeiro e das parcerias com a Universidade de São Paulo, Instituto Butantan, entre outros. O IBEx faz parte, em conjunto com a Fiocruz, UFRJ, UENF e outras Instituições de pesquisa, da Rede de Monitoramento de Dengue, Zika e Chikungunya no estado do Rio de Janeiro. Além dessas parcerias, o IBEx apoia tecnicamente o curso de Mestrado Profissional em Saúde, Medicina Laboratorial e Tecnologia Forense (MPSMLTF) da UERJ.
INTRODUÇÃO
Nas últimas três décadas, a incidência de infecções sistêmicas apresentou aumento significativo devido a fatores como Aids, transplantes de órgãos e medula óssea, uso de citostáticos e quimioterápicos, corticoterapia, antibioticoterapia, técnicas cirúrgicas invasivas e acesso vascular. Dentre os agentes fúngicos, Candida spp. é, indubitavelmente, o microrganismo mais envolvido em casos de infecção em pacientes imunocomprometidos. Além disso, essas leveduras têm se mostrado mais resistentes às drogas antifúngicas de uso clínico, principalmente quando espécies de Candida não albicans estão presentes. Importa mencionar que, de fato, qualquer organismo fúngico que apresente algum fator de patogenicidade poderá causar potencialmente infecção, a princípio, em indivíduos que pertençam a populações vulneráveis.(1,2)
As infecções fúngicas são a maior causa de morbimortalidade, principalmente em pacientes com malignidades hematológicas, sendo candidíases e aspergiloses os processos mais prevalentes (cerca de 90%).(3) Há consenso na literatura sobre o aumento na incidência das infecções fúngicas invasivas e alteração no espectro dos agentes etiológicos.(4)
CANDIDÍASE SISTÊMICA
Candidíases invasivas continuam liderando as causas de intercorrências fúngicas em pacientes imunocomprometidos em Unidades de Terapia Intensiva. A instituição de rápida e adequada terapia tem mostrado uma redução significativa nas taxas de morbidade e mortalidade e permitido a redução geral de custos hospitalares. Embora a Candida albicans permaneça sendo o principal agente responsável por infecções relacionadas à assistência à saúde, espécies de Candida não albicans, como o complexo C. glabrata, complexo C. parapsilosis, C. tropicalis, complexo C. guilliermondii, C. dubliniensis, C. lusitaniae e C. krusei têm emergido como significantes patógenos oportunistas. Dada a inerente variabilidade nos perfis de susceptibilidade antifúngica de diferentes espécies de Candida e a emergência de infecções por mais de uma espécie, a identificação correta da espécie é necessária para a melhor decisão terapêutica do clínico e conhecimento do comportamento epidemiológico.(5)
A candidíase sistêmica é a uma das formas micóticas mais polimórficas e, em muitos casos, se configura como um achado terminal. Comumente apresenta-se com sinais e sintomas inespecíficos, provas sorológicas inconclusivas e com a necessidade de equipamentos específicos para o isolamento e identificação do microrganismo.(6,7) Além disso, estão presentes altos percentuais de mortalidade e o custo de tratamento com frequência é elevado.(8)
Vários fatores são atribuídos ao risco de desenvolvimento de candidíase invasiva. A candidemia é observada, particularmente, entre pacientes hospitalizados por longos períodos de tempo, tendo como fatores predisponentes para fungemias, tratamentos com antibióticos de amplo espectro, quimioterapia, transplantes, uso de cateteres, nutrição parenteral, procedimentos invasivos múltiplos, neutropenia, pacientes imunossuprimidos e formação de biofilmes.(9,10)
O complexo Candida albicans ainda é considerado a espécie mais comum em candidemias, porém, o aumento das taxas de candidemia por Candida tropicalis, complexo Candida parapsilosis, complexo Candida glabrata e Candida krusei tem sido relatado em todo o mundo. A razão para emergência de espécies não albicans não está ainda completamente elucidada, mas algumas condições médicas podem produzir impacto no risco de desenvolvimento de candidemia por essas espécies. O complexo Candida parapsilosis é responsável por candidemias a partir do uso de cateteres e nutrição parenteral, Candida tropicalis está mais associada a neoplasias e neutropenias, e Candida krusei e complexo Candida glabrata estão mais associadas a candidemias de correntes de prévia exposição a azólicos.(8)
Diversas espécies de Candida isoladas em hemocultura demonstram sensibilidade à anfotericina B e ao fluconazol, porém, existem algumas exceções, como os isolados de Candida krusei, que são intrinsicamente resistentes ao fluconazol, e algumas do complexo Candida glabrata, que possuem sensibilidade reduzida a azólicos. A correta identificação da espécie e o teste de susceptibilidade são dados fundamentais para o adequado manejo das candidemias.(11)
IDENTIFICAÇÃO DE LEVEDURAS
Recentemente, as tecnologias para a identificação de leveduras têm avançado significativamente com métodos que incluem desde ensaios bioquímicos manuais e automatizados até ensaios baseados em análises de ácidos nucleicos e proteômica. Com o uso de métodos micológicos convencionais (tubo germinativo, produção de clamidósporos e meio cromogênico), a identificação de espécie de leveduras pode requerer de alguns dias a várias semanas. Alguns desses métodos, inclusive, não apresentam eficácia na identificação de espécies menos comuns. Embora as técnicas moleculares tenham aumentado consideravelmente a capacidade de identificação de leveduras, muitas delas estão associadas com custos elevados, complexidade de execução e necessidade de um conhecimento técnico específico. Diante desse quadro, a Espectrometria de Massas por Tempo de Voo (MALDI-TOF MS) se apresenta como uma alternativa promissora para a identificação rotineira de espécies de leveduras clínicas.(5)
Produção de Tubo Germinativo
O complexo Candida albicans tem a capacidade de produzir tubos germinativos na presença de soro (humano) ou meio hiperproteico, incubados à temperatura de 35ºC (±2ºC), por um período de 2-3 horas.(12) O teste é simples, eficiente e econômico para triagem e identificação de C. albicans. Aproximadamente, 95,97% dessas leveduras produzem tubo germinativo, contudo Candida dubliniensis e Candida tropicalis também são capazes de induzir crescimento de hifas nas mesmas condições que o complexo Candida albicans.(13)
Procedimento técnico:
- Inocular fragmentos de culturas de leveduras (após crescimento em ágar batata dextrosado – BDA, por
24-48 h) em 0,4 mL de soro humano, utilizando agulha ou alça bacteriológica;
- Incubar os tubos por um período de 2-3 h, a 35º ± 2;
- Colocar uma alíquota da suspensão entre lâmina e lamínula e examinar em microscópio com objetiva de
40x, procurando observar a produção de tubos germinativos.
Produção de Clamidósporos
Sob condições não ótimas de crescimento, Candida albicans pode desencadear a formação de clamidósporos, que são estruturas arredondadas e refringentes com uma parede celular espessa.(14)
Diversos meios de cultura são propostos para a indução de clamidósporos, sendo que os mais comuns possuem um baixo valor nutricional e altas taxas de carbono/nitrogênio. Para que haja a diminuição da tensão superficial, agentes como o Tween 80 são adicionados. O meio de ágar milho é o meio de eleição para a produção de clamidósporos.(12)
Procedimento técnico:
- Semear as colônias de leveduras através de estrias paralelas em ágar milho com Tween-80 (1%);
- Cobrir a superfície do meio com lamínulas estéreis, ficando estas sobre as estrias;
- Incubar à temperatura de 30º C, por 48-96 horas;
- Retirar as lamínulas, montá-las em lâminas e examinar em microscópio com objetiva de 40x, procurando observar a formação de clamidósporos.
Identificação Cromogênica
Essa identificação é procedida em um meio de cultura diferencia, que facilita o isolamento e a identificação presuntiva de algumas espécies de leveduras clinicamente importantes, a partir da coloração de suas colônias.(15)
Substratos cromogênicos são adicionados como constituintes no meio, o que, com o desenvolvimento das leveduras, determina o surgimento de colônias de cores características para cada espécie (Figura 1).

Figura 1. Meio cromogênico CHROMágar® – Candida.
A utilidade do meio cromogênico, contudo, é limitada, pois esses meios são capazes de discriminar um número restrito de leveduras (complexo Candida albicans, Candida tropicalis e Candida krusei) e também porque múltiplas espécies de leveduras podem desenvolver cores semelhantes, afetando assim, a especificidade.(16)
O meio permite a identificação do complexo Candida albicans pela coloração verde produzida, Candida tropicalis pela coloração azul petróleo e Candida krusei pela coloração rosa e textura seca.(17) Outras espécies de Candida desenvolvem coloração entre branca e malva.
Procedimento técnico:
- Semear os isolados de leveduras em meio cromogênico;
- Incubar a 35ºC ±2, por 24 horas;
- Observar a cor exibida pelas colônias e identificar segundo orientações do fabricante.
Análise por Bioquímica Automatizada: Vitek 2
O Vitek 2 é um sistema automatizado correntemente utilizado em rotinas de laboratórios de microbiologia clínica. O equipamento realiza automaticamente os processos necessários para a identificação bioquímica de microrganismos e determinação da susceptibilidade a antimicrobianos, utilizando um inóculo primário isolado de culturas positivas. Embora os métodos de identificação clássicos, manuais e morfológicos ainda sejam considerados padrão ouro, esses métodos são lentos, laboriosos e são propensos a interpretações subjetivas. Por outro lado, o sistema Vitek 2 reduz o tempo necessário para identificação e permite a padronização dos resultados inter e intra laboratórios, armazenamento de resultados, emissão de relatórios epidemiológicos rápidos e testes simultâneos de susceptibilidade a antimicrobianos.(18)
Esse sistema utiliza cartões com substratos bioquímicos liofilizados para a identificação taxonômica, a partir da assimilação de carboidratos e nitratos, e também permite a determinação do perfil de susceptibilidade de leveduras.(19)
O equipamento apresenta um banco de dados que tem sido progressivamente atualizado, para abranger leveduras comuns e infrequentes de espécies clínicas.(20)
Procedimento Técnico:
- Utilizar cartões para identificação de leveduras (YST);
- Inocular os cartões segundo instruções do fabricante;
- Inserir no módulo leitor para leitura automatizada dos substratos;
- Considerar as identificações com percentuais de probabilidade ³ a 85%.
- Realizar testes complementares, quando necessário.
Análise Proteômica: Espectrometria de Massas por Tempo de Voo – [Maldi Tof Ms]
A espectrometria de massas em sua configuração MALDI-TOF (Matrix Assisted Laser Desorption/Ionization Time of Flight) é um método analítico desenvolvido em meados de 1988, que evoluiu rapidamente para espectros de impressão digital para vários microrganismos, incluindo bactérias e fungos, através da análise de perfis de proteínas.
Bancos de dados foram posteriormente desenvolvidos e adotados em laboratórios de microbiologia clínica. Esses bancos de dados empregam o conceito de reconhecimento de padrões ou de impressão digital, onde o espectro de massa obtido de uma bactéria ou levedura é comparado com espectros existentes para encontrar a correspondência mais próxima.(21)
Nesse sistema, os agentes microbianos são colocados em uma placa com uma matriz constituída de ácido a-Ciano-4-hidroxi-cinamico (HCCA). A placa é irradiada com um laser que vaporiza a amostra (dessorção), ionizando as moléculas, que serão conduzidas por uma diferença de potencial elétrico, a uma região livre de campos elétricos, no tubo de voo. Nessa região, ocorre a distinção dos íons de acordo com as suas velocidades, que são inversamente proporcionais às respectivas relações massa/carga. Os íons são detectados, após sua chegada ao final do tubo, com registro do seu tempo de voo, associando-se esse parâmetro indiretamente com a massa molecular da proteína.(22,23,24)
A abordagem do biomarcador para a identificação de microrganismos emprega proteínas específicas encontradas dentro da célula. Proteínas ribossomais tornaram-se um dos biomarcadores ideais porque são abundantes, altamente conservadas e codificadas por genes cromossômicos. Essas proteínas têm massas moleculares que se enquadram de 4 KDa a 30 KDa.(21)
Um obstáculo para a identificação de isolados de leveduras, usando MALDI-TOF MS, é a presença de uma robusta parede celular que inibe a análise direta de leveduras. Pesquisadores têm superado essa dificuldade usando uma variedade de abordagens de pré-processamentos destinados a liberar as proteínas intracelulares, que são a base para a identificação por MALDI-TOF MS.(16)
O MALDI-TOF MS se apresenta como uma alternativa real para a identificação rotineira de espécies de leveduras. É um sistema que providencia a correta identificação de espécies, levando apenas alguns minutos para executar todo o processo, sendo relativamente barato para conduzir a identificação específica.(5)
Procedimento Técnico:
- Aplicar as colônias de leveduras isoladas diretamente na placa do equipamento;
- Tratar com 1 µL de ácido fórmico a 85%. Após a secagem em temperatura ambiente, aplicar 1 µL da matriz HCCA e submeter à secagem também à temperatura ambiente.(25,26)
- Inserir a placa, posteriormente, no equipamento que irá irradiar com laser, que vaporizará a amostra, ionizando as moléculas que serão então aspiradas e elevadas ao detector (Figura 2);
- Os dados obtidos através do perfil do espectro serão comparados com o banco de dados do equipamento, utilizando o software do fabricante para a identificação dos microrganismos;
- Analisar as amostras em duplicata no Microflex LT MALDI-TOF MS da Bruker Daltonik, Biotyper (Fremont, CA), no modo linear positivo, em uma frequência 60 Hz, com um intervalo de massa entre 2 KDa e 20 KDa (Figura 3);
- As informações obtidas serão analisadas automaticamente, usando o software Bruker Biotyper 3.1. Cada espectro será obtido a partir de 240 tiros, em passos de 40 tiros de diferentes posições da placa alvo, e o log dos scores serão calculados;
- A identificação do ponto de corte dos scores será interpretada do seguinte modo: scores ³ 1,7 indicarão identificação confiável a nível de espécie, enquanto que scores <1,7 indicarão identificação não confiável, requerendo a repetição da análise.(26)
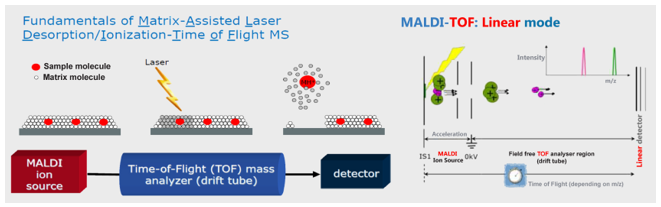
Figura 2. Fundamento da metodologia de Espectrometria de Massas por Tempo de Voo – Microflex LT Maldi-Tof MS da Bruker Daltonik, Biotyper (Fremont, CA). Fonte: Manual da Bruker.

Figura 3. Equipamento de Espectrometria de Massas por Tempo de Voo, Microflex LT Maldi-Tof MS da Bruker Daltonik, Biotyper (Fremont, CA) – IBEx. Fonte: IBEx.
Análise Molecular: PCRem Tempo Real
O PCR em Tempo Real combina a química com uma sonda fluorescente, para detecção do produto amplificado, na mesma reação.
A avaliação da fluorescência é obtida pela plataforma do equipamento em cada ciclo de amplificação. Em geral, a reação ocorre em menos de uma hora, sendo consideravelmente mais rápida que as técnicas de PCR convencionais.(27)
Este tipo de detecção do amplicon constitui um avanço significativo na química dos ácidos nucleicos, por ser mais rápido que os métodos tradicionais de detecção de amplicons. Além disso, há uma redução significativa na chance de contaminação do amplicon no laboratório, pois não há abertura do tubo de reação para a análise do amplicon. Existem diferentes plataformas disponíveis para a amplificação de ácidos nucleicos em tempo real.(28)
Várias são as moléculas fluorogênicas que podem ser usadas para a detecção dos ácidos nucleicos amplificados, numa reação homogênea ou em tempo real. Essas moléculas podem ser inespecíficas e detectar qualquer tipo de ácido nucleico amplificado (SYBR Green) ou específicas, o que significa que elas são oligonucleotídeos e se hibridizam a uma sequência específica, presente no amplicon (sondas).(29)
O SYBR Green é um corante que se liga ao sulco menor do DNA de dupla hélice. Emite muito pouca fluorescência, quando no estado não ligado, mas gera uma fluorescência consideravelmente maior quando ligado ao DNA, essa propriedade o torna útil para a determinação da presença de um produto de DNA amplificado.(29)
Procedimentos técnicos
- Semear todas as amostras em BDA e incubar a 35°C±2°C, por 18-24 h
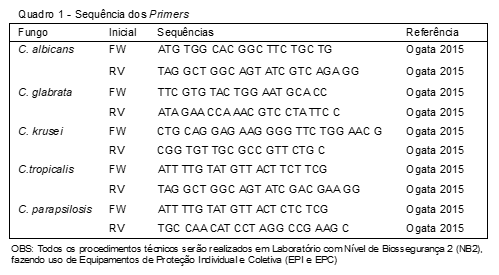
- Para extração do DNA, preparar suspensões em 20 mL de solução de lise (SDS 10% 125µL; NaOH 0,05N 100µL; H2O qsp 5000µL). Estas suspensões deverão ser aquecidas a 94ºC, por 5 min e, posteriormente, adicionadas de 180 µL de água Mili-Q estéril e centrifugadas a 16.000 g, por 5 min. Uma alíquota de 3 mL do DNA extraído em suspensão deverá ser adicionada juntamente com 1mL (10pmol) de cada um dos dois iniciadores específicos e 5mL de PCR Luminaris Hi-Green qPCR Master Mix (Thermo Scientific, MA, USA), em um volume total de 10 mL para a reação de PCR em Tempo Real O desenho dos Primers para o PCR é sumarizado no Quadro 1.
- Toda a mistura deverá ser submetida a 1 ciclo de 95°C, por 5 min, para a desnaturação do DNA, 30 ciclos de 95°C, por 30s, 60°C, por 30s, e 72°C, por 30s, 1 ciclo de 72°C, por 7 min e, posteriormente, manter a 4°C em termociclador em tempo real (StepOnePlus, Applied Biosystems– Thermo Fisher).
COLEÇÃO DE CULTURA DE MICRORGANISMOS
Representando desde pequenos centros privados a grandes centros de serviços e possuindo diferentes objetivos, políticas e acervos, as coleções de cultura são importantes instituições de preservação de recursos biológicos. Essas coleções estão frequentemente associadas às atividades de uma organização parental, como instituições acadêmicas ou de pesquisa científica, e mantêm organismos que podem ser utilizados de formas distintas.(30)
Coleções de cultura de microrganismos são centros de conservação da biodiversidade, sendo encarregadas de coletar e disponibilizar organismos relevantes para a pesquisa científica, bem como estão ligadas ao desenvolvimento tecnológico e ao ensino. A preservação e a manutenção de culturas devem ser realizadas de forma a garantir a sobrevivência do microrganismo, a estabilidade das propriedades morfológicas e fisiológicas, características genéticas e a pureza dos isolados durante períodos prolongados.(31)
Existem diversos tipos de coleções microbiológicas ex-situ que são classificadas de acordo com o atendimento prestado. As coleções de trabalho são aquelas mantidas nos laboratórios e utilizadas em linhas específicas de pesquisa (podendo ser extintas quando os pesquisadores mudam de linha); coleção institucional, que abastece diversas linhas de pesquisa de uma única instituição; coleções de serviço, que possuem acervo abrangente, curadoria profissional e têm papel fundamental na aquisição, caracterização taxonômica e tecnológica, manutenção e distribuição de microrganismos para toda a comunidade para fins industriais ou de pesquisa.(32)
Subcultivos periódicos e sucessivos de isolados fúngicos podem acarretar o aparecimento de alterações morfológicas, fisiológicas e bioquímicas, além do elevado risco de contaminação. Desse modo, diversas coleções de cultura têm utilizado metodologias de preservação de longo prazo, mas não há um método universal para todos os tipos de fungos.(33-36)
MÉTODO DE PRESERVAÇÃO: CONSERVAÇÃO E ESTOCAGEM DE LEVEDURAS EM ÁGUA
DESTILADA ESTÉRIL
A importância da manutenção e preservação de microrganismos, caracteriza-se como reflexo da necessidade de utilização desses recursos a qualquer momento, quer para fins experimentais, didáticos, industriais ou estudos comparativos.(37) Dessa forma, definir a técnica de preservação de culturas mais adequada e dispor de procedimentos simples e eficientes, reveste-se de grande valia aos laboratórios de microbiologia.(38)
Diversos métodos vêm sendo empregados para preservação de fungos, porém, em virtude da diversidade biológica desses microrganismos, não existe uma técnica padrão que seja capaz de preservá-los de forma única ou generalizada.(39)
A escolha do método de manutenção mais adequado deve ser baseada nas características do agente em estudo, assim como nas vantagens e desvantagens de cada técnica.(38,39) O alvo de qualquer método de manutenção é preservar a viabilidade e, principalmente, proporcionar estabilidade genética ao microrganismo isolado pelo maior tempo possível, evitando assim a formação excessiva de mutações que alterem suas características.(40,41)
Diferentes métodos têm sido propostos para a manutenção de fungos em coleções cultura, como preservação em óleo mineral estéril, congelamento, congelamento em temperatura ultra baixa, água destilada estéril (Método de Castellani) e liofilização.(34) O método de Castellani é um método simples e economicamente viável.(42-45)
O método de Castellani (Figura 4) consiste no armazenamento de microrganismos em água esterilizada ou solução salina, sendo indicado na preservação de microrganismos sensíveis a baixas pressões osmóticas de soluções hipotônicas.(46-48) O método se baseia na transferência de culturas para frascos contendo uma solução de água destilada esterilizada, com posterior armazenamento sob temperatura ambiente. As suspensões de células deverão ser bem concentradas, a partir de um crescimento em meio sólido, ou poderão ser introduzidos blocos de ágar contendo os microrganismos. Um pequeno espaço físico deverá ser disponibilizado para o acondicionamento dos frascos.(38)

Figura 4. Cepas de leveduras mantidas pelo método de Castellani. Fonte: IBEx.
Segundo McGinnis, Padhye & Ajello,(34) o método de manutenção de cultura de fungos em água destilada esterilizada, por longos períodos de tempo, é simples, econômico e confiável. O método oferece muitas vantagens para laboratórios que mantêm pequenas coleções de cultura para referência ou propósitos de ensino. O espaço de armazenamento necessário para os frascos é mínimo, e como os frascos são armazenados à temperatura ambiente a refrigeração não se faz necessária. A técnica de revitalização é menos laboriosa que as culturas preservadas em óleo mineral estéril.
Procedimento Técnico:
- Inocular fragmentos das colônias, assim como fragmentos do meio de cultura em tubos estéreis, contendo 5 mL de água destilada esterilizada;
- Manter os tubos à temperatura ambiente e sob refrigeração entre 2 a 8ºC.
BOAS PRÁTICAS DE COLEÇÃO DE CULTURA DE LEVEDURAS DO IBEx
Essas práticas constituem um conjunto de normas, procedimentos e atitudes de segurança, que visam a minimização dos acidentes que envolvem as atividades laboratoriais dentro da Coleção de Cultura do IBEx. (19,49)
Equipamentos de Segurança Laboratorial
São considerados barreiras primárias de contenção, que visam a proteção dos profissionais.(50)
- Equipamentos de Proteção Individual (EPI): Dispositivos destinados a proteger a saúde e a integridade física do profissional,(51)representado por:
o Jaleco de manga longa;
o Luvas descartáveis;
o Luva térmica;
o Óculos de proteção;
o Máscara descartável;
o Calçado fechado;
o Pro-pé.
- Equipamentos de Proteção Coletiva (EPC): permitem a execução de operações seguras para o operador e demais profissionais do laboratório:(50)
o Cabine de segurança biológica classe II B2;
o Cabine de segurança biológica classe III – “Glove Box”;
o Autoclave;
o Chuveiro e lava olhos;
o Pipetador automático;
o Extintores de incêndio;
o Caixa coletora de pérfuro cortante;
o Sistema de controle de acesso com identificação biométrica.
Biossegurança na Coleção de Cultura de Leveduras do IBEx
Conceitos
- Bioconfiança (biosurety): É o conjunto de sistemas e procedimentos para salvaguardar os agentes biológicos e toxinas contra furto, roubo, perda, desvio, acesso ou uso não autorizado e garantir que todas essas ações sejam conduzidas de maneira segura e confiável, englobando nesse conceito a biossegurança, a bioproteção e os controles de pessoal e material;(52)
- Biossegurança (biosafety): É o conjunto de ações destinadas a prevenir, controlar, reduzir ou eliminar riscos inerentes às atividades que possam, de forma não intencional, comprometer a saúde humana, animal, vegetal e o ambiente;(52)
- Bioproteção (biossecurity): É o conjunto de ações que visam a minimizar o risco do uso indevido, roubo e/ou a liberação intencional de material com potencial risco à saúde humana, animal, vegetal e ambiental.(52)
Principais normas de biossegurança:
- Utilização adequada de EPIs e EPCs;
- Lavagens das mãos antes e após os procedimentos técnicos;
- Descontaminação das bancadas antes e após as análises;
- Manusear, transportar e armazenar materiais biológicos e microrganismos de forma segura;
- Acondicionar os resíduos biológicos de forma adequada (saco branco leitoso);
- Descontaminar por autoclavação os resíduos biológicos e demais materiais contaminados;
- Obedecer a sinalização de risco do laboratório;
- Seguir as normas institucionais de treinamentos e segurança laboratorial.
Abstract
Objective: The Compendium of Methods and Good Practices in Yeast Culture Collection of the Brazilian Army Biology Institute (IBEx) was elaborated with the purpose of operationalizing the research activities, guaranteeing the safe and reliable performance of its regimental objectives. Methods: The IBEx Yeast Culture Collection was created from isolated strains of systemic candidiasis, through the use of manual and automated methodologies to authenticate them, in order to guarantee scientific research and teaching activities. Results: The Institute adapted and modernized its research laboratories by creating the Center for Biodefense Studies, with Biosafety Level NB-3. Based on the need for organization to meet the demands of biological defense, the IBEx has been improving its structure to dominate and guarantee new technologies of identification and management of the microorganisms involved in Bioterrorism. Conclusion: The compilation of methodologies in the form of a Compendium provided the operationalization of the IBEx Culture Collection.
Keywords
Yeasts; blood culture; clinical diagnosis; research and new techniques
REFERÊNCIAS
- Dornelas-Ribeiro M, Pinheiro EO, Guerra C, Braga-Silva LA, Carvalho SMF, Santos ALS, et al. Cellular characterisation of Candida tropicalis presenting fluconazole-related trailing growth. Mem. Inst. Oswaldo Cruz [Internet]. 2012;107(1):31-38. Available from: http://www. scielo.br/scielo.php?script=sci_arttext&pid=S0074-02762012000100005&lng=en.
- Neufeld PM, Melhem Mde S, Szeszs MW, Ribeiro MD, Amorim Ede L, da Silva M, Lazéra Mdos S. Nosocomial candidiasis in Rio de Janeiro State: distribution and fluconazole susceptibility profile. Braz J Microbiol. 2015;46(2):477-84.
- Ruhnke M, Maschmeyer G. Management of mycosis in patients with hematologic disease and cancer – review of the literature. Eur J Med Res. 2002 May 31;7(5):227-35.
- Neufeld PM. Caracterização taxonômica e susceptibilidade a antifúngicos de leveduras isoladas de infecção hospitalar. Tese de Doutorado. INCQS/Fiocruz. 2009.
- Chao QT, Lee TF, Teng SH, Peng LY, Chen PH, Teng LJ, Hsueh PR. Comparison of the accuracy of two conventional phenotypic methods and two MALDI-TOF MS systems with that of DNA sequencing analysis for correctly identifying clinically encountered yeasts. PLoS One. 2014;9(10):e109376.
- Londero AT, Wanke B, Lazera MS, Monteiro PCF. Micologia Médica, Fiocruz, Revisão. 2004.
- Figueras C, Díaz de Heredia C, García JJ, Navarro M, Ruiz-Contreras J, Rossich R, et al; Grupo de Estudio de la Infección Fúngica Invasiva de la Sociedad Española de Infectología Pediátrica (SEIP). The Spanish Society of Paediatric Infectious Diseases (SEIP) recommendations on the diagnosis and management of invasive candidiasis. An Pediatr (Barc). 2011 May;74(5):337.e1-337.e17. doi: 10.1016/j.anpedi.2010.12.012. [Article in Spanish]
- Colombo AL, Nucci M, Park BJ, Nouér SA, Arthington-Skaggs B, da Matta DA, et al.; Brazilian Network Candidemia Study. Epidemiology of candidemia in Brazil: a nationwide sentinel surveillance of candidemia in eleven medical centers. J Clin Microbiol. 2006;44(8):2816-23.
- Tumbarello M, Posteraro B, Trecarichi EM, Fiori B, Rossi M, Porta R, et al. Biofilm production by Candida species and inadequate antifungal therapy as predictors of mortality for patients with candidemia. J Clin Microbiol. 2007;45(6):1843-50.
- Vigouruoux S. Morin O, Moreau P, Harousseau JL, Milpied N. Candidemia in patients with hematologic malignancies: analysis of 7 years experience in a single center. Haematologica, v. 91, n 5, p. 717-718. 2006.
- Sandven P. Epidemiology of candidemia. Rev Iberoam Micol 2000; 17: 73-81.
- Dornelas-Ribeiro M. Caracterização do efeito “trailing” em cepas de Candida tropicalis e sua influência na morfologia, ultraestrutura e expressão de aspartil peptidases. Tese de Doutorado. IMPPG-UFRJ, Rio de Janeiro, Brasil. 83p. 2011.
- Campanha NH, Neppelenbroek KH, Spolidorio DM, Spolidorio LC, Pavarina AC. Phenotypic methods and commercial systems for the discrimination between C. albicans and C. dubliniensis. Oral Dis. 2005 Nov;11(6):392-8.
- Lee KH, Shin WS, Kim D, Koh CM. The presumptive identification of Candida albicans with germ tub induced by high temperature. Yonsei Med J. 1999 Oct;40(5):420-4.
- Odds FC, Bernaerts R. CHROMagar Candida, a new differential isolation medium for presumptive identification of clinically important Candida species. J Clin Microbiol. 1994;32(8):1923-9.
- Buchan BW, Ledeboer NA. Advances in identification of clinical yeast isolates by use of matrix-assisted laser desorption ionization-time of flight mass spectrometry. J Clin Microbiol. 2013;51(5):1359-66. doi: 10.1128/JCM.03105-12.
- Ribeiro PM, Ito CYC, Junqueira JC, Jorge AOC. Isolamento de Candida spp. com utilização de meio de cultura cromogênico CHROMagar Candida. Braz Dent Sci 2009 out./dez.;12 (4):40-45. DOI: https://doi.org/10.14295/bds.2009.v12i4.641
- Monteiro AC, Fortaleza CM, Ferreira AM, Cavalcante Rde S, Mondelli AL, Bagagli E, da Cunha Mde L. Comparison of methods for the identification of microorganisms isolated from blood cultures. Ann Clin Microbiol Antimicrob. 2016 ;15(1):45. DOI 10.1186/s12941-016-0158-9. 2016.
- Araujo CR, Miranda KC, Passos XS, Souza LKH, Lemos J A, Abbas Khrais CH, et al. Identificação de Leveduras do Gênero Candida por Métodos Manuais convencionais e pelo método Cromogênico CHROMagarTM-Candida. Rev. Patol. Trop.. 2005;34(1):37-42.
- Posteraro B, Efromov L, Leoncini E, Amore R, Posteraro P, Ricciardi W, Sanguinetti M. Are the conventional commercial yeast identification methods still helpful in the era of new clinical microbiology diagnostics? A meta-analysis of their accuracy. J Clin Microbiol. 2015;53(8):2439-50. doi: 10.1128/JCM.00802-15.
- Kassim A, Pflüger V, Premji Zul, Daubenberger C, Revathi G. Comparison of biomarker based Matrix Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry (MALDI-TOF MS) and conventional methods in the identification of clinically relevant bacteria and yeast. BMC Microbiol. 2017;17(1):128. doi: 10.1186/s12866-017-1037-z.
- Karas M, Hillenkamp F. Laser desorption ionization of proteins with molecular masses exceeding 10,000 daltons. Anal Chem. 1988 Oct 15;60(20):2299-301.
- Gross JH. Mass spectrometry: a textbook. Springer Science & Business Media. 2006.
- Denis J, Machouart M, Morio F, Sabou M, Kauffmann-Lacroix C, Contet-Audonneau N, et al. Performance of Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry for Identifying Clinical Malassezia Isolates. J Clin Microbiol. 2016;28;55(1):90-96. doi: 10.1128/JCM.01763-16.
- Bulane A, Hoosen A. Use of matrix-assisted laser desorption/ionisation – time of flight mass spectrometry analyser in a diagnostic microbiology laboratory in a developing country. Afr J Lab Med. 2017; 6(1):598. https://doi.org/10.4102/ajlm.v6i1.598. 2017.
- Lee HS, Shin JH, Choi MJ, Won EJ, Kee SJ, Kim SH, Shin MG, Suh SP. Comparison of the Bruker Biotyper and Vitek MS Matrix-Assisted Laser Desorption/Ionization Time-of-Flight mass spectrometry systems using a formic acid extraction method to identify common an uncommon yeast isolates. Ann Lab Med. 2017 May;37(3):223-230. doi: 10.3343/alm.2017.37.3.223.
- Espy MJ, Uhl JR, Sloan LM, Buckwalter SP, Jones MF, Vetter EA, et al. Real-time PCR in clinical microbiology: applications for routine laboratory testing. Clin Microbiol Rev. 2006;19(1):165-256. Erratum in Clin Microbiol Rev. 2006 Jul;19(3):595.
- Foy CA, Parkes HC. Emerging homogeneous DNA-based technologies in the clinical laboratory. Clin Chem. 2001;47(6):990-1000.
- Koneman EW, Winn WC, Allen SD, Janda WM, Procop GW, Schreckenberger PC, Woods GL. Koneman Diagnóstico Microbiológico, texto e atlas colorido. Guanabara Koogan. 6ª edição. 2008. 1.565 p
- Consenso. Recomendações para operação e gerenciamento de coleções de culturas de microrganismos. Sociedade Brasileira de Microbiologia. 2006.
- Cavalcanti SDB. Aplicação de metodologias de preservação e caracterização de fungos na coleção de culturas do Instituto de Medicina Tropical de São Paulo. Dissertação de Mestrado em Ciências da Faculdade de Medicina da USP. 2010.
- Andrade TS, Bastos LT, Scola MCG, Felippe JMM. Seção de coleção de culturas do Instituto Adolfo Lutz – 68 anos de história dedicados à saúde pública. Bepa 2008;5(59):10-15.
- Butterfield W, Jong SC, Alexander MT. Preservation of living fungi pathogenic for man and animals. Can. J. Microbiol. 1974;20:1665-1673. https://doi.org/10.1139/m74-258
- Mcginnis MR, Padhye AA, Ajello L. Storage of stock cultures of filamentous fungi, yeasts, and some aerobic actinomycetes in sterile distilled water. Appl Microbiol. 1974 Aug; 28(2): 218-222.
- Qiangqiang Z, Jiajun W, Li L. Storage of fungi using sterile distilled water or lyophilization: Comparison after 12 years. Mycoses. 1998 May-Jun;41(5-6):255-7.
- Cavalcante SC, Freitas RS, Vidal MS, Dantas DC, Levi JE, Martins JEC. Evaluation of phenotypic and genotypic alterations induced by long periods of subculturing of Cryptococcus neoformans strains. Mem. Inst. Oswaldo Cruz [Internet]. 2007;102(1):41-47. Disponível em: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0074-02762007000100006&lng=pt.
37.Guimarães LC. Métodos de preservação de fungos potencialmente toxigênicos. Lavras, MG. Universidade Federal de Lavras – UFLA. 2011.
- Sola MC, Oliveira AP, Feistel JC, Rezende CSM. Manutenção de microrganismos: conservação e viabilidade. Enciclopédia biosfera. Goiânia. 8 (14):1398-1418. 2012.
- Dellaretti EM. Preservação de fungos em baixas temperaturas. Sete Lagoas, MG. Universidade Federal de São João. UFSJ. 2014.
- Abreu MMV, Tutunji VL. Implantação e manutenção da coleção de culturas de microorganismos do UniCEUB. Universitas: Ciências da Saúde, Brasília, v.02 n.2, p. 236-25, 2004.
- Girão MD, Prado MR, Brilhante RSN, Cordeiro RA, Monteiro AJ, Sidrim JJC, Rocha MFG. Viabilidade de cepas de Malassezia pachydermatis mantidas em diferentes métodos de conservação. Rev. Soc. Bras. Med. Trop. 2004;37(3):229-233. Available from: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0037-86822004000300007&lng=en
- Rodrigues EG, Lírio VS, Lacaz CS. Preservação de fungos e actinomicetos de interesse médico em água destilada. Rev. Inst. Med. trop. S. Paulo [online]. 1992, vol.34, n.2, pp.159-165. Available from: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0036-46651992000200012&lng=en.
- Figueiredo MB. Métodos de preservação de fungos patogênicos. Biológico. v. 63, n. 1/2, p. 73-82. 2001.
- Canhos VP, Umino CY, Manfio GP. Coleções de culturas de microrganismos. Resumo: Coleções de culturas de microrganismos. Fundação de Amparo à Pesquisa do Estado de São Paulo, FAPESP – Centro de Referência em Informação Ambiental – CRIA, 2004.
- Nakasone KK, Peterson AW, Jong S. Preservation and distribution of fungal cultures. In: Mueller G M, Bills G F, Foster M S. Biodiversity of fungi, inventory and monitoring methods. 1ª ed. San Diego: Elsevier Academic Press. p. 37-47. 2004.
- Pimentel CPV, Figueiredo MB. Métodos de preservação de fungos em meio de cultura. Biológico, São Paulo, 55 (1/2): 27-33. 1989.
- Costa CP, Ferreira MC. Preservação de microrganismos: revisão. Revista de Microbiologia, São Paulo, 22 (3): 263-268. 1991.
- Neufeld PM, Oliveira PC. Preservação de dermatófitos pela técnica da água destilada estéril. Revista Brasileira de Análises Clínicas, São Paulo, 40 (3):167-169. 2008.
- Mastroeni MF. Biossegurança Aplicada a Laboratórios e Serviços de Saúde. 2ª ed. Atheneu. 2005, 338 p.
- Hirata MH, Hirata RDC, Mancini Filho J. Manual de Biossegurança. Editora Manole. 2ª edição. 2012.
- Portaria GM N.º 3.214, NR 6 – Dispõe sobre Equipamento De Proteção Individual – Epi, De 08 De Junho De 1978. 06/07/78.
- Portaria Normativa Nº 585/MD, Diretrizes de Biossegurança, Bioproteção e Defesa Biológica do Ministério da Defesa de 7 de março de 2013.
Correspondência
Marcos Dornelas Ribeiro
Rua Francisco Manuel, 102, Benfica
Rio de Janeiro-RJ, Brasil
