Perfil de resistência a antimicrobianos de Enterobacteriaceae isoladas de secreção traqueal e hemocultura de pacientes em uma Unidade de Terapia Intensiva
Drug resistance profile of Enterobacteriaceae isolated from tracheal secretion and blood cultures of patients in an Intensive Care Unit
Carlos Alberto Medeiros Neto1
Maria Izabely Silva Pimentel2
Yasmin Alves Gomes1
Igor Vasconcelos Rocha3
Sibele de Oliveira Ribeiro4
1Graduando de Biomedicina no Centro Universitário Tabosa de Almeida. Caruaru-PE, Brasil.
2Centro Universitário Tabosa de Almeida. Caruaru-PE, Brasil.
3Biomédico, Doutorando. Instituto Aggeu Magalhães. Recife-PE, Brasil.
4Biomédica, mestre em Biotecnologia, doutora em Ciências Biológicas, docente da Asces-Unita. Caruaru-PE, Brasil.
Instituição: Centro Universitário Tabosa de Almeida. Caruaru-PE, Brasil.
Conflitos de interesse: sem conflitos de interesse.
Suporte financeiro: todo suporte financeiro foi de responsabilidade dos autores.
Recebido em 28/11/2019
Aprovado em 24/04/2020
DOI: 10.21877/2448-3877.202000927
INTRODUÇÃO
Pacientes de uma Unidade de Terapia Intensiva (UTI) apresentam estados críticos de saúde e encontram-se mais susceptíveis a adquirir infecções relacionadas à assistência à saúde (IRAS). Estas infecções possuem caracteres multifatoriais, sejam eles intrínsecos, como a saúde do paciente, ou extrínsecos, como o tempo de internação. Dentro do ambiente hospitalar, a UTI é o local que mais apresenta taxas de IRAS, consequentemente também apresenta os maiores índices de mortalidade. Amostras destes pacientes apresentam um alto padrão de resistência, principalmente urina, secreção traqueal e sangue, além dos procedimentos invasivos que contribuem significativamente para o aumento dos índices de infecção hospitalar. Como principais agentes etiológicos envolvidos nas IRAS, destacam-se as bactérias Gram negativas multirresistentes, sendo fundamental a identificação laboratorial destes como uma maneira de contribuir no direcionamento da antibioticoterapia.(1-4)
A secreção traqueal é um dos principais sítios de isolamento de microrganismos e trata-se da amostra biológica mais utilizada para o diagnóstico das infecções relacionadas com o trato respiratório, apresentando grande positividade para bactérias Gram negativas do grupo das enterobactérias.(5)
As hemoculturas são os exames realizados para observação da presença de microrganismos na corrente sanguínea e tratam-se de uma forma de diagnóstico de alta sensibilidade e especificidade, sugerindo, quando positivas, a disseminação de um processo infeccioso.(3,6) Silva et al.(7) destacam o isolamento de bactérias Gram positivas nos quadros de bacteremia, entretanto, quando ocorre identificação de Gram negativas têm-se a prevalência das enterobactérias e podem apresentar um quadro de multirresistência aos antimicrobianos com significativa piora do quadro clínico dos pacientes.
As enterobactérias estão comumente relacionadas nas IRAS, destacando-se: Escherichia coli, Klebsiella spp., Proteus mirabilis, Enterobacter spp., Salmonella spp., Citrobacter spp., predominando Escherichia coli, Klebsiella spp. e Enterobacter spp..(8)
A resistência antimicrobiana dessa família é um grande problema de saúde pública, sendo um fato que aumenta o tempo de internação dentro das UTIs.(5,9) Segundo o Clinical and Laboratory Standards Institute (CLSI), a resistência aos antimicrobianos é um fenômeno relacionado à formação de isolados de microrganismos que são capazes de se desenvolver na presença de elevada concentração antimicrobiana. Há vários mecanismos de resistência envolvidos, destacando-se a produção de b-lactamases de espectro estendido (Extended-Spectrum Beta-Lactamase, ESBL) que tem sido o mais comum e importante mecanismo de resistência das enterobactérias, além da produção de carbapenemases e resistência à polimixina B.(10)
Neste contexto, objetivou-se verificar a resistência das enterobactérias aos antimicrobianos dispostos pelo CLSI, isoladas de pacientes de UTI de um hospital do Agreste Pernambucano, juntamente com a análise de seus principais mecanismos de resistência.
MATERIAL E MÉTODOS
Trata-se de um estudo observacional, descritivo e transversal onde foram avaliadas hemocultura e secreção traqueal dos pacientes em uma Unidade de Terapia Intensiva de um hospital de Caruaru-PE, no período de agosto de 2018 a setembro de 2019. As amostras de secreção traqueal e hemocultura foram selecionadas por conveniência e obtidas pelos profissionais de saúde do hospital de estudo e encaminhadas, sob condições adequadas no que se refere à biossegurança, para o Laboratório de Microbiologia da Asces-Unita.
As hemoculturas foram semeadas utilizando-se uma seringa de 3 mL, sendo retirados cerca de 0,25 mL de sangue do frasco de hemocultura e realizado o semeio por esgotamento em Agar MacConkey. A secreção traqueal foi entregue em frascos estéreis denominados “bronquinho”, e em seguida semeada também em Agar MacConkey, com incubação a 37ºC durante 18-24 horas.(11)
Foi realizada coloração de Gram para observação e comprovação da morfologia bacteriana, e, após, realizou-se semeio nos testes bioquímicos Agar Triple Sugar Iron (TSI) para observação do metabolismo bacteriano frente aos açúcares contidos no meio para identificação laboratorial fenotípica, Ágar Citrato Simmons para observação do consumo de carbono, Ágar SIM (Sulfito, Indol e Motilidade) e Ágar Ureia.(12)
O teste para análise do perfil de resistência aos microbianos foi realizado a partir do método de disco-difusão de Kirby-Bauer em Agar Mueller Hinton, conforme proposto pelo CLSI. Foram utilizados os seguintes antibióticos: azitromicina (AZI), aztreonam (ATM), cefepime (CPM), cefotaxima (CTX), ciprofloxacina (CIP) e gentamicina (GEN).
O teste para verificação da produção de ESBL foi realizado pelo método de disco-difusão, sendo utilizadas cefalosporinas de 1ª e 3ª gerações e um inibidor de beta-lactamase, amoxicilina + ácido clavulânico (AMC), cefepime (CPM), ceftazidima (CAZ) e ceftriaxona (CRO). A AMC foi colocada centralmente no meio Agar Mueller Hinton e os outros discos foram posicionados a uma distância de aproximadamente 25 mm, com as placas sendo incubadas a 37°C por 18-24 horas. É considerado produtor de ESBL quando há formação de um halo ao redor do disco de amoxicilina + ácido clavulânico.(13)
Para se verificar a produção de Klebsiella pneumoniae carbapenemase (KPC) foi realizado o teste de Hodge, preparando-se uma suspensão de Escherichia coli (ATCC 25922) em soro fisiológico, ajustada à escala 0,5 de MacFarland. Preparou-se uma diluição de 1:10 em soro fisiológico, e, em seguida, com ajuda de um swab inoculou-se em uma placa de Agar Mueller Hinton. Foi inserido um disco de imipenem no centro da placa e em seguida realizou-se semeio com a amostra suspeita, sendo a placa incubada a 37ºC por 18 a 24 horas.(14) A confirmação de KPC foi verificada pela presença de uma deformidade no halo de inibição bacteriana.(13)
Ética
O presente estudo foi desenvolvido após a aprovação do hospital de estudo por meio da carta de anuência assinada pelo diretor do mesmo e aprovação pelo Comitê de Ética em Pesquisa do Centro Universitário Tabosa de Almeida (Asces-Unita) sob o número de Parecer nº 3.200.710.
RESULTADOS
De 58 amostras clínicas utilizadas no estudo (49 de hemocultura e nove de secreção traqueal), todas apresentaram crescimento bacteriano, sendo vinte (34%) Gram negativas que foram utilizadas no estudo. Destas, 17 (85%) foram referentes a enterobactérias, sendo nove (53%) oriundas de secreção traqueal e oito (47%) de hemocultura. Em tais amostras foram identificadas Proteus mirabilis (41%), Serratia rubidaea (17%), Enterobacter aerogenes (12%), Enterobacter sakazaki (6%), Providencia rettgeri (6%), Klebsiella oxytoca (6%), Klebsiella pneumoniae (6%) e Escherichia coli (6%), conforme esquematizado na Figura 1.
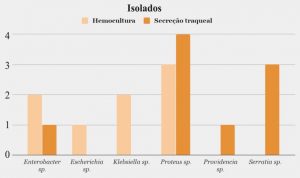 Figura 1. Principais enterobactérias isoladas no presente estudo na Unidade de Terapia Intensiva.
Figura 1. Principais enterobactérias isoladas no presente estudo na Unidade de Terapia Intensiva.
Analisando-se o perfil de resistência, foi verificado que 65% dos isolados tiveram resistência a azitromicina e aztreonam, 76% a cefepime e cefotaxima, 70% a ciprofloxacina e 35% a gentamicina.
Dos isolados de Serratia spp., 33% apresentaram resistência a azitromicina e ciprofloxacina e 67% a aztreonam, cefotaxima e cefepime.
Setenta e um por cento das bactérias do gênero Proteus spp. encontradas mostraram resistência frente a azitromicina, cefepime, cefotaxima e ciprofloxacina, 57% a aztreonam e 43% a gentamicina, sendo duas delas resistentes a todos os antibióticos testados.
Quanto às cepas de Enterobacter spp., 68% revelaram resistência a gentamicina e 100% a azitromicina, aztreonam, cefepime, cefotaxima e ciprofloxacina. E a Providencia spp. encontrada mostrou resistência a aztreonam, cefepime e cefotaxima.
Das espécies de Klebsiella spp. encontradas, todas mostraram 100% de resistência à maioria dos antibióticos, exceto à gentamicina, em que essa porcentagem foi de 50%. O único isolado de Escherichia spp. apresentou resistência apenas à ciprofloxacina.
Em relação à produção de ESBL, 35% dos isolados apresentaram positividade para a mesma, sendo três Proteus mirabilis, duas Enterobacter aerogenes e uma Klebsiella pneumoniae, todas oriundas de hemocultura. Pode-se observar o halo ao redor do disco de AMC, indicando produção da enzima beta-lactamase (Figura 2).
Quarenta e um por cento dos isolados apresentaram positividade para produção da enzima KPC, sendo cinco Proteus mirabilis, um Serratia rubidae e um Klebsiella oxytoca. Observou-se a deformidade no halo formado ao redor do disco de IPM (Figura 3).
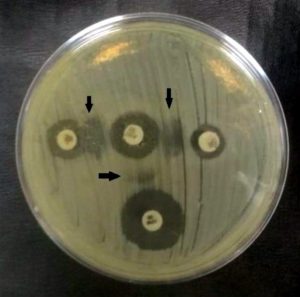
Figura 2. Positividade para produção de ESBL. Fonte: autores
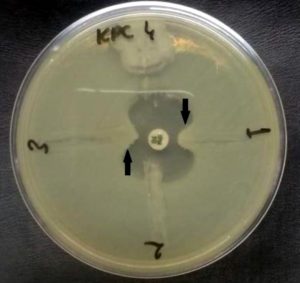 Figura 3. Positividade para produção de KPC. Fonte: autores
Figura 3. Positividade para produção de KPC. Fonte: autores
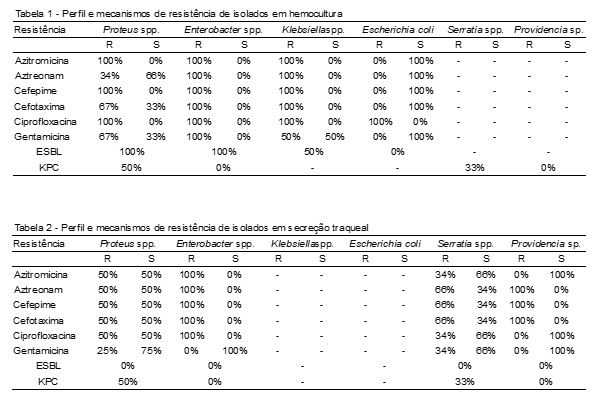
As Tabelas 1 e 2 separam os gêneros bacterianos, o perfil e o mecanismo de resistência em cada sítio de isolamento.
DISCUSSÃO
A ordem Enterobacterales está presente em várias situações, podendo ser encontrada na microbiota intestinal ou como causadora de infecções severas com alta capacidade de resistência aos antimicrobianos. Dentro do ambiente hospitalar são comumente isoladas em secreção traqueal, hemocultura, urocultura e escarro.(15,16)
Carvalho e Gontijo Filho(17) e Paredes(18) mostram que, na UTI, das bactérias Gram negativas isoladas, prevaleceram as da família Enterobacteriaceae, corroborando com o presente estudo, que, de 20 isolados Gram negativos, 17 foram enterobactérias, com um representativo de 85%.
A hemocultura é um tipo de amostra altamente encontrada na UTI e está relacionada a bacteremia de alta letalidade.(15) Magalhães et al.(16) observaram em seu estudo um crescimento bacteriano de 22% nas amostras de hemocultura, sendo contrário ao presente estudo, que obteve 100% de crescimento bacteriano. No nosso estudo, dentro das cepas oriundas de hemocultura, o gênero mais isolado foi Proteus spp., tendo outras pesquisas na literatura que observaram o mesmo.(19-21) Houve outros bacilos isolados que estão frequentemente relacionados a altas taxas de infecções hospitalares, como Enterobacter spp. e Escherichia spp., conforme outros estudos.(16,19) É perceptível que não são tão citados na literatura quando os relacionam à hemocultura.
Em relação a secreção traqueal, Carvalho et al.(22) relataram um crescimento bacteriano de 59% das amostras em sua pesquisa, em contraste com os 100% de crescimento no presente estudo. Debarba et al.(5) e Carvalho et al.(22) alegaram que a prevalência dos achados são de Gram negativos, sendo observado também no nosso estudo, onde 100% dos achados foram Gram negativos. De acordo com Serafim et al.,(23) o predomínio de Gram negativos nessas amostras advém de sua versatilidade nutricional, a qual propõe uma sobrevida no ambiente hospitalar, sendo o objetivo do presente estudo.
Souza,(24) em sua pesquisa de Prevalência de microrganismos em secreção traqueal em pacientes da UTI de hospital público, mostrou que, de 108 amostras, 10,3% foram do gênero Enterobacter spp., 13,4% de Serratia spp. e 23,1% de Proteus spp., apresentando resultados semelhantes ao presente estudo, com maior presença de Proteus spp. (23,5% dos isolados). Em relação a Escherichia spp. e Klebsiella spp., o presente estudo não teve nenhum isolado desses gêneros nessa amostra clínica, o que não aconteceu na pesquisa já citada,(24) que teve um percentual de 0,9% de ambos os gêneros.
Paredes,(18) em sua Pesquisa de enterobactérias oriundas na UTI, obteve 92,3% dos isolados derivados de secreção traqueal, prevalecendo em relação aos isolados do sangue, o que aconteceu também no presente estudo, que apresentou 52,9% dos isolados em secreção traqueal.
Caracterizada como um problema de saúde pública, a resistência microbiana está altamente relacionada à morbidade, mortalidade e elevação dos custos durante a assistência, o que colabora com o agravo das infecções nas unidades de terapia intensiva.(15)
Os isolados obtiveram maior resistência à classe das cefalosporinas de 3ª e 4ª gerações (76,4% resistentes à cefepime e cefotaxima), indo de encontro à literatura.(25) Mota et al.,(21) em sua pesquisa de Perfil e prevalência de resistência aos antimicrobianos de bactérias Gram-negativas isoladas de pacientes de uma unidade de terapia intensiva, mostrou grande resistência a quinolonas (88,8%), assemelhando-se à presente pesquisa, que foi a segunda classe em que os isolados tiveram maior resistência, representativo de 70,5%.
No estudo de Basso et al.,(26) o isolado Proteus spp. obteve 100% de resistência aos antibióticos cefotaxima e ciprofloxacina, prevalecendo em relação ao presente estudo, em que as bactérias desse gênero isolados de hemocultura apresentaram taxa de resistência de 100% e 67% aos antibióticos citados, respectivamente, e de 50% para ambos os antibióticos nos isolados de secreção traqueal.
Em pesquisa do Perfil microbiológico relacionado à assistência à saúde em uma unidade de terapia intensiva em um hospital da Zona da Mata mineira,(27) em amostras de secreção traqueal, hemocultura e outras, dois isolados de Enterobacter spp. foram 100% resistentes a cefepime e ciprofloxacina, corroborando com a presente pesquisa. Além disso, também a alguns antibióticos da classe de cefalosporinas de 3ª geração e beta-lactâmicos, que no presente estudo foram utilizados cefotaxima e aztreonam, respectivamente.
No presente estudo, o gênero Escherichia spp. obteve 100% de resistência à ciprofloxacina, tendo resultados semelhantes em um estudo do Peru,(18) onde 85,1% dos isolados de Escherichia spp. tiveram resistência a esse antibiótico. O mesmo corroborou com o presente estudo, onde o gênero Klebsiella spp. obteve 50% de sensibilidade à gentamicina. Escherichia spp. e Klebsiella spp. estão entre as quatro bactérias classificadas como prioridade crítica de resistência.(18)
De forma geral, os isolados de Serratia spp. e Providencia spp. dessa pesquisa foram encontrados apenas na secreção traqueal e mostraram uma alta taxa de resistência a aztreonam, cefepime e cefotaxima.
A resistência bacteriana tem vários mecanismos, na família Enterobacteriaceae o mais importante tem sido a produção de betalactamases do tipo ESBL e KPC.(28)
No presente estudo, todos os isolados produtores de ESBL foram oriundos de hemocultura. Silva et al.,(7) em sua pesquisa de frequência de betalactamase de espectro estendido (ESBL) em isolados de hemoculturas, observaram 77,33% de produtores de ESBL no universo de trinta amostras de Gram negativos, assemelhando-se com o nosso estudo, que detectou 75% de produtores dessa enzima nas amostras de hemocultura. Neste estudo foram encontrados quatro gêneros bacterianos, sendo dois deles, Proteus spp. e Enterobacter spp., também isolados no trabalho de Silva et al.(7) Todos os isolados de ESBL foram altamente resistentes aos antibióticos testados.
Paula et al.(29) relatam que, na literatura, as taxas de Proteus spp. oriundos de secreção traqueal e sangue produtores de carbapenemases são poucas, porém, quando encontradas, é de suma preocupação. No presente trabalho, esse gênero bacteriano foi o isolado que mais apresentou positividade para a produção de KPC, com um representativo de 30%. Um isolado de Serratia spp. e um de Klebsiella spp. também foram produtores desta enzima.
CONCLUSÃO
As Enterobactérias encontradas neste estudo apresentaram grande resistência aos antibióticos testados. Esses microrganismos mostraram-se produtores de ESBL e KPC, que são os mecanismos de resistência mais envolvidos nessa família, alguns até mostrando produção simultânea dessas duas enzimas, trazendo então uma preocupação maior em relação aos mesmos. Estudos como este podem ajudar o profissional a adquirir uma antibioticoterapia mais eficaz para o paciente.
Agradecimentos
Ao laboratório de microbiologia do Hospital Regional do Agreste, que nos disponibilizou as amostras.
Abstract
Objective: Check the presence and resistance profile of enterobacteria against antimicrobials, from tracheal secretion and blood culture from an Intensive Care Unit (ICU). Metods: Mac Conkey Agar, biochemical tests and Gram were used for bacterial identification. Antimicrobial resistance was identified by the disc diffusion method and the disc approach and modified Hodge tests were performed to verify the production of ESBL and KPC, respectively. Results: Nine isolates from tracheal secretion and eight from blood culture were identified: Proteus mirabilis (41%), Serratia rubidaea (17%), Enterobacter aerogenes (12%), Enterobacter sakazaki (6%), Providencia rettgeri (6%), Klebsiella oxytoca (6%), Klebsiella pneumoniae (6%) and Escherichia coli (6%). The antimicrobial resistance test showed multiresistant isolates: 35.3% were producers of ESBL and 41.1% of KPC. Conclusion: The identification of these multiresistant pathogens their resistance mechanisms is very important for effective treatment and faster recovery for patients.
Keywords
Enterobacteriaceae; blood culture; Intensive Care Unit; drug resistance
REFERÊNCIAS
- Souza MAS, Nascimento GC, Bim FL, Oliveira LB, Oliveira ADS. Infecções hospitalares relacionadas a procedimentos invasivos em unidades de terapia intensiva: revisão integrativa. Rev Pre Infec e Saúde. 2017;3(3):49-58. Disponível em: http://www.ojs. ufpi.br/index.php/nupcis/article/download/4251/pdf.
- Ruschel BD, Rodrigues DA, Formolo F. Perfil de resultados de hemoculturas positivas e fatores associados. RBAC. 2017;49(2): 158-63. Disponível em: http://www.rbac.org.br/wp-content/uploads/2017/08/RBAC-vol-49-2-2017-ref.-503-finalizado.pdf.
- Leão LSNO, Passos XS, Reis C, Valadão LMA, Silva MRR, Pimenta FC. Fenotipagem de bactérias isoladas em hemoculturas de pacientes críticos. Rev Soc Bras Med Trop. 2007;40(5):537-40. Disponível em: http://www.scielo.br/pdf/rsbmt/v40n5/a09v40n5.pdf.
- Costa FJMD. Resistência à Polimixina B em bactérias gram-negativas carbapenemas resistentes isoladas em hospitais do Rio Grande do Norte. Natal. Dissertação [Mestrado em Ciências Biológicas] – Centro de Biociências, Universidade Federal do Rio Grande do Norte; 2017. Disponível em: https://repositorio.ufrn.br/jspui/handle/123456789/23159
- Debarba E, Silvero KSV, Teixeira JJV, Silva CM, Peder LD. Prevalência microbiana em secreções traqueais de pacientes em unidade de terapia intensiva – Experiência de 4 anos. JIC. 2018;7(1). Disponível em: http://www.jic-abih.com.br/index.php/jic/article/view/188/pdf
- Araújo MRE. Hemocultura: recomendações de coleta, processamento e interpretação dos resultados. J Infect Control. 2012;1(1):08-19. Disponível em: http://www.iqg.com.br/pbsp/img_up/01355393320.pdf
- Silva RCG, Silva AC, Oliveira SR. Resistência microbiana e frequência de betalactamase de espectro estendido (ESBL) em isolados de hemoculturas. J Bras Patol Med Lab. 2014 dez;50(6):421-7. Disponível em: http://www.scielo.br/pdf/jbpml/v50n6/1676-2444-jbpml-50-06-0421.pdf
- Lavagnoli LS, Bassetti BR, Kaiser TDL, Kutz KM, Junior CC. Fatores associados à aquisição de Enterobactérias resistentes aos carbapenêmicos. Rev. Latino-Am. Enfermagem. 2017;25:e2935. Disponível em: http://www.scielo.br/pdf/rlae/v25/pt_0104-1169-rlae-25-e2935.pdf
- Marques JB, et al. Molecular characterization of Enterobacteriaceae resistant to carbapenem antimicrobials. J Bras Patol Med Lab. 2015;5(3):162-5. Disponível em: http://www.scielo.br/pdf/jbpml/v51n3/1676-2444-jbpml-51-03-0162.pdf
- Santos JG, Fonseca BO, Bello AR, Pereira JAA. Avaliação de transferência de marcadores de resistência por conjugação em enterobactérias resistentes aos antimicrobianos e telurito de potássio no ambiente hospitalar. Revista Pensar. 2018;4:30-41. Disponível em: http://revistapensar.com.br/biologia/pasta_upload/artigos/a159.pdf
- Oliveira MEF, Araújo DG, Oliveira SR. Resistance of non-fermenting Gram-negative bacilli isolated from blood cultures from an emergency hospital. J Bras Patol Med Lab. 2017 Apr;53(2):87-91. Disponível em: http://www.scielo.br/scielo.php?script=sci_arttext&pid =S1676-24442017000200087&lng=en
- Tortora GJ, Funke BR, Case CL. Microbiologia. 12thed. Porto Alegre: ARTMED; 2017.
- Organização Pan-Americana da Saúde. Boas Práticas em Microbiologia Clínica. 1aed. são Paulo: Unifesp; 2008.
- Laborclin Produtos para Laboratórios Ltda. Manual de Antibiograma. Rev 16. 2019 Mar. Disponível em: https://www.laborclin.com.br/wp-content/uploads/2019/05/Manual_antibiograma_2019.pdf
- Basso ME, Pulcinelli RSR, Aquino ARC, Santos KF. Prevalência de infecções bacterianas em pacientes internados em uma unidade de terapia intensiva (UTI). RBAC. 2016;48(4):383-8. Disponível em: http://www.rbac.org.br/wp-content/uploads/2017/04/RBAC-vol-48-4-2016-ref.-307.pdf
- Magalhães LS, Abreu ES, Pussente CG, Oliveira CGA. Incidência e perfil de sensibilidade e resistência das estirpes bacterianas isoladas das hemoculturas de um hospital oncológico. Revista Científica da FAMINAS. 2014 Mai-Ago;10(2):39-53. Disponível em: http://www.faminas.edu.br/upload/downloads/20141126155759_473711.pdf
- Carvalho RH, Gontijo Filho PP. Epidemiologically relevant antimicrobial resistance phenotypes in pathogens isolated from critically ill patients in a Brazilian university hospital. Braz. J. Microbiol. 2008;39:623-30. Disponível em: http://www. scielo.br/pdf/bjm/v39n4/arq05.pdf
- Paredes LYR. Resistencia antimicrobiana de enterobacterias en pacientes de unidade de cuidados intensivos. Trujillo. Tese [Título de médico cirurgião] – Facultad de Medicina, Universidad Privada Antenor Orrego; 2015. Disponível em: http://repositorio.upao.edu.pe/ bitstream/upaorep/1604/1/Rodriguez_Paredes_ Antimicrobiana_Enterobacterias_Cuidados.pdf
- Dallacorte TS, Indras DM, Teixeira JJV, Peder LD, Silva CM. Prevalência e perfil de sensibilidade antimicrobiana de bactérias isoladas de hemoculturas realizadas em hospitais particulares. Rev Inst Adolfo Lutz. 2016;75:1702. Disponível em: http://www.ial.sp.gov.br/ resources/insituto-adolfo-lutz/publicacoes/rial/10/rial75_completa/artigos-separados/1702.pdf
- Freire ILS, Araujo RO, Vasconcelos QLDAQ, Menezes LCC, Costa IKF, Torres GF. Perfil microbiológico, de sensibilidade e resistência bacteriana das hemoculturas de unidade de terapia intensiva pediátrica. Rev Enferm UFSM. 2013 Sep-Dec;3(3):429-39. Disponível em: https://periodicos.ufsm.br/reufsm/article/view/8980/pdf.
- Mota FS, Oliveira HA, Souto RCF. Perfil e prevalência de resistência aos antimicrobianos de bactérias Gram-negativas isoladas de pacientes de uma unidade de terapia intensiva. RBAC. 2018;50(3):270-7. Disponível em: http://www.rbac.org. br/wp-content/uploads/ 2019/01/RBAC-vol-50-3-2018-ref-740-final.pdf.
- Carvalho MAP, Meneses LBA, Souto RADM, Barros MAA, Moraes MN, Cordeiro RC. Revista de Enfermagem, UFPE Online. 2013 set;7(9):5366-742013. Disponível em: https://periodicos.ufpe.br/revistas/revistaenfermagem/article/download/11818/14215
- Serafim SD, Souza JA, Soares JC, Forno NLF. Coleta de secreção traqueal: estudo comparativo de técnicas. Saúde (Santa Maria). 2015 Jan-Jul;41(1):57-64. Disponível em: https://periodicos.ufsm.br/revistasaude/article/view/11376/pdf.
- Souza GM. Prevalência de microrganismos em secreção traqueal em pacientes da UTI de hospital público. 2011 Nov. Disponível em: https://www.webartigos.com/artigos/prevalencia-de-microrganismos-em-secrecao-traqueal-em-pacientes-da-uti-de-hospital-publico/80302
- Furtado DMF, Silveira VS, Carneiro ICRS, Furtado DMF, Kilishek MP. Consumo de antimicrobianos e o impacto na resistência bacteriana em um hospital público do estado do Pará, Brasil, de 2012 a 2016. Rev Pan Amaz Saude. 2019;10. Disponivel em: http://scielo.iec.gov.br/pdf/rpas/v10/2176-6223-rpas-10-e201900041.pdf.
- Basso ME, Pulcinelli RSR, Aquino ARC, Santos KF. Prevalência de infecções bacterianas em pacientes internados em uma unidade de terapia intensiva (UTI). RBAC. 2016;48(4):383-388. Disponível em: http://www.rbac.org.br/wp-content/uploads/2017/04/RBAC-vol-48-4-2016-ref.-307.pdf.
- Correa MEG, Fidelis CF, Valadares FD, Neto JAR, Ribeiro VSS, Soares CQG. Perfil microbiológico relacionado à assistência à saúde em uma unidade de terapia intensiva em um hospital da Zona da mata Mineira. Revis Científica Fagoc Saúde. 2018;3(1):49-58. Disponível em: https://revista.fagoc.br/index.php/saude/article/view/340/305.
- Oliveira CBS, Dantas VCB, Neto RM, Azevedo PRM, Melo MCN. . Frequência e perfil de resistência de Klebsiella spp. em um hospital universitário de Natal/RN durante 10 anos. Rev Bras Patol Med Lab. 2011;47(6):589-594. Disponível em: https://www.redalyc.org/ pdf/3935/393541963003.pdf.
- Paula VG, Quintanilha LV, Silva FAC, Rocha HF, Santos FL. Enterobactérias produtoras de carbapenemase: prevenção da disseminação de superbactérias em UTI’s*. Universitas: Ciências da Saúde. 2016 Jul-Dez;14(2):175-185. Disponível em: https://www. publicacoesacademicas.uniceub.br/cienciasaude/article/download/3847/3276.
Correspondência
Carlos Alberto Medeiros Neto
Centro Universitário Tabosa de Almeida
Av. Portugal, 584 – Universitário
55016-400 – Caruaru-PE, Brasil
