Prevalência e perfil de resistência de bactérias isoladas de hemoculturas de pacientes atendidos em um hospital de referência cardiológica de Pernambuco
Prevalence and resistance profile of bacteria isolated from blood cultures of patients attended in a cardiological reference hospital in Pernambuco
Vera Lúcia Nascimento Bezerra1
Wêndeo Kennedy Costa2
Edgar Brito de Souza Júnior3
Marismar Fernandes do Nascimento4
Luciana Rezende Bandeira de Melo4
Andrea Maria André Gomes1
Veridiana Câmara Furtado5
1Farmacêutica. Centro Universitário Maurício de Nassau (UNINASSAU). Aracaju-SE, Brasil.
2Doutorando. Universidade Federal de Pernambuco (UFPE). Recife-PE, Brasil.
3Biomédico. Universidade de Pernambuco (UPE). Recife-PE, Brasil.
4Universidade de Pernambuco (UPE). Recife-PE, Brasil.
5Centro Universitário Maurício de Nassau (UNINASSAU). Aracaju-SE, Brasil.
Instituição: Universidade de Pernambuco, Pronto Socorro Cardiológico (PROCAPE), Recife – PE, Brasil.
Conflitos de interesse: os autores declaram não haver conflitos de interesse.
Recebido em 05/11/2020
Aprovado em 14/01/2021
DOI: 10.21877/2448-3877.202001980
INTRODUÇÃO
A sepse é a resposta inflamatória sistêmica a um quadro infeccioso confirmado. Qualquer foco de infecção pode evoluir para sepse, que representa um sério problema de saúde mundial, estando frequentemente associada a um aumento considerável nas taxas de morbidade e mortalidade. No Brasil, a taxa de mortalidade é em torno de 65%, enquanto que a média mundial é de 30%, segundo dados do Instituto Latino Americano de Sepse (ILAS).(1-3)
Aproximadamente 40% dos pacientes em sepse apresentam resultados positivos para presença de microrganismos na hemocultura, já pacientes em choque séptico tem esse número elevado para aproximadamente 70%. Essa situação ocorre por diversos fatores como: tempo de internamento, doença primária, procedimentos invasivos, infecções recorrentes e terapia prolongada por uso de antibióticos.(4)
Quando um processo infeccioso se instala na corrente sanguínea, a detecção do agente patogênico ocorre exclusivamente por meio de exames laboratoriais e o resultado contribui para a determinação da conduta terapêutica que deverá ser tomada para combater o agente.(5)
Entre os microrganismos que predominam nas hemoculturas destacam-se as bactérias Gram-positivas, como Staphylococcus coagulase negativa (SCoN), S. aureus e Enterococcus spp. Já entre as bactérias Gram-negativas destacam-se Acinetobacter baumannii, Klebsiella pneumoniae, Pseudomonas aeruginosa e Enterobacter spp.(6,7)
O tratamento da sepse deve ser realizado utilizando-se antibioticoterapia, que apresenta uma ação sobre os microrganismos. O uso do antibiótico adequado permite uma terapia mais eficaz, de acordo com o perfil de sensibilidade do microrganismo, reduzindo o risco de aparecimento de bactérias resistentes. Porém, o uso indiscriminado e descontrolado de antibióticos pode favorecer o desenvolvimento de bactérias multidrogas resistentes, que constitui um sério problema de saúde pública mundial.(8,9)
Entre as bactérias preocupantes pelo desenvolvimento de alto grau de resistência antimicrobiana, destacam-se o S. aureus resistente à metilicina (MRSA), Staphylococcus coagulase negativa resistente à metilicina (MRSCoN), Enterococcus sp. vancomicina resistentes (VRE), P. aeruginosa, Acinetobacter spp. e espécies da família Enterobacteriaceae produtoras de b-lactamases de espectro ampliado (ESBL), e produtoras de carbapenemases do tipo Klebsiella pneumoniae carbapenemase (KPC). Esta situação submete o paciente a terapia antimicrobiana combinada, que muitas vezes não é eficaz no tratamento, permitindo que o paciente chegue ao óbito.(10,11)
Diante do exposto, é indispensável a realização de um levantamento do perfil bacteriano nos pacientes hospitalizados com suspeita de septcemia, as quais podem ser causadas por bactérias que apresentem elevado perfil de resistência. Os resultados poderão contribuir para medidas de controle da disseminação e da resistência dos microrganismos, bem como auxiliar no uso de antibióticos para tratamento eficaz no combate das bactérias que apresentam grau de resistência, diminuindo assim as taxas de morbidade e mortalidade de pacientes no ambiente hospitalar.
Material e Métodos
Foi realizado um estudo retrospectivo referente ao período de janeiro a dezembro de 2017, baseado em dados microbiológicos obtidos dos registros de Hemocultura do Laboratório de Análises Clínicas do Pronto Socorro Cardiológico de Pernambuco – Prof. Luiz Tavares (Procape), pertencente ao complexo hospitalar da Universidade de Pernambuco (UPE). Este hospital presta assistência aos pacientes de todas as regiões Norte/Nordeste com atendimento de emergência de média e alta complexidade cardiovascular, englobando desde assistência ambulatorial a cirurgias cardíacas 24 horas.
As amostras clínicas procedentes de todas as enfermarias foram analisadas de acordo com a sua procedência, mês e dados bacteriológicos. Foram excluídos do estudo amostras de hemocultura de líquidos pericárdico, pleural e ascíticos.
As amostras de hemoculturas foram processadas em equipamento automatizados BacT/Alert® 3D sistemas de detecção microbiana e Vitek 2 compact da Biomerieux®, no qual se realiza a identificação microbiológica do gênero e espécie através de cartão de identificação individual com código de barras, composto de 64 poços com provas bioquímicas liofilizadas, individuais sem necessidade de adição de reagentes reveladores. E cartões com 18 antibióticos, para realizar teste de antibiograma com antibióticos dos grupos: aminoglicosídeos, carbapenêmicos, b-lactâmicos, quinolonas, sulfonamidas e glicopeptídeos. Os resultados obtidos foram armazenados no Observa versão 04.03 da Biomerieux®. Em seguida foram transferidos para uma planilha do Excel 2017 contendo informações sobre as bactérias identificadas e o perfil do antibiograma.
Ética
O presente estudo teve aprovação do comitê de ética em pesquisa com seres humanos da Universidade de Pernambuco sob o CAAE: 11860919.4.0000.5207, número do Parecer: 3.321.540.
RESULTADOS
No período compreendido entre janeiro a dezembro de 2017 foram analisadas 3.323 amostras de hemoculturas dos pacientes hospitalizados nas unidades de terapia intensiva e enfermarias. Destas amostras, 3.203 (96,38%) apresentaram resultado negativo e 120 (3,62%) apresentaram resultado positivo para bactérias. Destas, 71 (59,16%) bactérias eram Gram-positivas e 49 (40,84%) Gram-negativas. A presença de culturas mistas, que indicavam a presença de mais de um microrganismo, não foi observada.
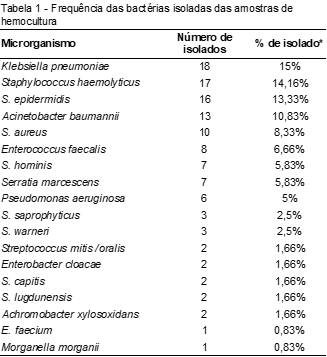
Foram identificadas 19 espécies bacterianas nas hemoculturas, sendo os microrganismos mais frequentes K. pneumoniae (15%), seguido de S. haemolyticus (14,16%) e S. epidermidis (13,33%). S. haemolyticus e S. epidermidis juntas com outras espécies de Staphylococcus diferentes de S. aureus correspondem ao grupo Staphylococcus coagulase negativa, que apresentou um total de 50 isolados (41,66%).
Dos 120 isolados, 19 apresentaram perfil de sensibilidade e 101 mostraram perfil de resistência a um ou mais de um grupo de antibióticos (Tabela 2). Dentre os grupos de antibióticos utilizados para combater bactérias, aqueles aos quais as bactérias demonstraram maior porcentagem de resistência foram b-lactâmicos (67,5%), quinolonas (43,33) e carbapenêmicos (21,6).
Várias bactérias apresentaram perfil de multirresistência, como E. cloacae (2) que os dois isolados apresentaram resistência aos grupos de antimicrobianos: b-lactâmicos, quinolonas e carbapenêmicos e um isolado também apresentou resistência aos aminoglicosídeos. A. baumannii (13) apresentou dez isolados também resistentes aos b-lactâmicos, quinolonas e carbapenêmicos.
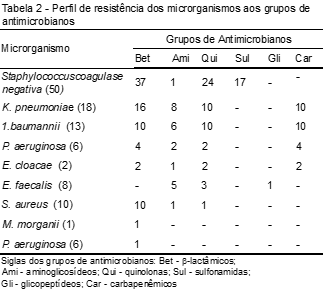
- pneumoniae (18) apresentou 16 isolados resistentes aos b-lactâmicos e dez isolados resistentes aos carbapenêmicos e quinolonas. Já P. aeruginosa (6) apresentou quatro isolados com resistência a dois grupos de antimicrobianos: b-lactâmicos e carbapenêmicos e dois isolados com resistência aos aminoglicosídeos e quinolonas.
Dos dez isolados de S. aureus todos apresentaram resistência aos Beta-lactâmicos e um isolado com resistência aos aminoglicosídeos e quinolonas. O grupo Staphylococcus coagulase negativa (50) apresentou perfil de resistência a quatro grupos de antibióticos, 37 aos b-lactâmicos, 24 aos quinolonas, 17 aos sulfonamidas e apenas um isolado apresentou resistência aos aminoglicosídeos.
DISCUSSÃO
A prevalência de bactérias resistentes aos antimicrobianos em ambiente hospitalar faz com que o tratamento não seja efetivo, prologando o processo de internação hospitalar e muitas vezes levando o paciente ao óbito. O diagnóstico se torna de extrema importância para o controle da morbidade e mortalidade dos pacientes, como também para controlar possíveis infecções hospitalares. Esse diagnóstico é obtido através das análises de hemoculturas, que podem ser realizadas por métodos manuais ou automatizados, sendo esse último mais eficiente por apresentar os resultados mais rápidos e com menos trabalho técnico.(12,13)
No presente estudo, bactérias Gram-positivas apresentaram prevalência maior que as Gram-negativas, essa prevalência deve-se principalmente à presença de S. aureus e Staphylococcus coagulase negativa concordando com outros estudos. No estudo realizado por Sousa et al.,(5) que avaliou a prevalência de hemoculturas positivas de paciente em UTI de um hospital universitário, a prevalência de Gram-positivas foi de 52,35%. O mesmo foi observado no estudo realizado por Meneghetti et al.,(14) que apresentou prevalência de 53,92% e também avaliou hemoculturas positivas em um hospital universitário.
As bactérias do grupo Staphylococcus coagulase negativa foram os microrganismos com maior prevalência. Este grupo de bactérias se apresenta como principal etiologia dos quadros de hemoculturas positivas nos hospitais brasileiros, sendo também encontrados em outros estudos com alta prevalência, como no estudo realizado por Ruschel,(15) que avaliou os resultados de hemoculturas positivas de um hospital no Rio Grande do Sul e que de 150 isolados 45,60% relatavam Staphylococcus coagulase negativa. O mesmo foi observado por Santana et al.,(16) em um hospital terciário que dos 113 isolados 60,10% representavam Staphylococcus coagulase negativa.
- pneumoniae foi a segunda bactéria com maior prevalência no presente estudo, apresentando 18 isolados. O mesmo pode ser observado nos estudos realizados por Sousa et al.,(17) onde K. pneumoniae apresentou prevalência de 10,7% (10). Já nos estudos realizados por Sousa et al.,(5) e Ruschel, (15) apesar de não ser o microrganismo com maior predominância, ainda assim foi encontrado elevado número de isolados de K. pneumoniae nas hemoculturas, com relatados de prevalência de 14 (8,4%)e 9 (6,0%), respectivamente.
A produção da enzima Klebsiella pneumoniae carbapenemase (KPC) é um agravante problema associado à espécie K. pneumoniae, pois tornam-se resistentes a grupos de antibióticos como os b-lactâmicos e aminoglicosídeos, sendo um dos principais desafios quando se fala em resistência microbiana, tendo em vista que a KPC apresenta uma taxa de mortalidade de aproximadamente 50%. Além de ser encontrada em isolados de K. pneumoniae, a enzima KPC também pode ser encontrada em Enterobacter sp.(18,19)
Outro mecanismo associado a K. pneumoniae é a capacidade de produção de enzimas hidrolíticas denominadas beta-lactamases de espectro ampliado (ESBL), que apresentam a capacidade de inativar os b-lactâmicos. Além de ser comumente encontrada em K. pneumoniae e E. coli, ESBL também pode ser encontrada em menor intensidade em P. aeruginosa e A. baumannii.(20,21)
No presente estudo, os isolados de E. cloacae apresentaram 100% de resistência a três grupos de antibióticos: b-lactâmicos, quinolonas e carbapenêmicos. Já isolados de K. pneumoniae apresentaram perfil de resistência de aproximadamente 89% aos b-lactâmicos e de aproximadamente 56% aos quinolonas e carbapenêmicos. Para os aminoglicosídeos, esse índice de resistência foi de aproximadamente 45%. Multirresistências em K. pneumoniae também foram relatados em outros trabalhos, como no de Moura Pinto et al.,(22) e Alves,(23) onde os isolados apresentaram resistências de 68% e 62% a um ou mais grupo de antibióticos, respectivamente.
Embora A. baumannii não tenha sido a bactéria mais prevalente entre os isolados, ela apresentou resistência de 76,92% a três grupos de antibióticos: b-lactâmicos, carbapenêmicos e quinolonas. Elevada resistência vem sendo encontrada em isolados de A. baumannii, inclusive frente a antimicrobianos de amplo espectro, como pode ser observado nos estudos realizados por Sousa et al.,(5) e Queiroz et al.(24)
Podem ser encontrados em P. aeruginosa diferentes mecanismos de resistência, esses mecanismos reduzem a sensibilidade aos antimicrobianos gerando mecanismos de resistência cruzada e elevando as taxas de mortalidade, como exemplo a capacidade de produção de ESBL.(24,25) No presente estudo, P. aeruginosa (6) apresentou 66,67% de resistência a dois grupos de antimicrobianos: b-lactâmicos e carbapenêmicos e 33,33% apresentaram resistência aos aminoglicosídeos e quinolonas. Alta incidência de P. aeruginosa multidroga resistentes também foram encontradas no estudo realizado por Pires et al.(26)
Os Staphylococcus com resistência à meticilina/oxacilina situam-se entre os agentes de infecção hospitalar mais frequentes. Esta resistência ocorre devido a alterações genéticas que reduzem a ação de antimicrobianos b-lactâmicos.(22) S.aureus apresentou 100% de resistência aos b-lactâmicos, já Staphylococcus coagulase negativas isoladas mostraram de resistência de 74% aos b-lactâmicos. Foram encontradas Staphylococcus coagulase negativa com 43,90% de resistência a meticilina,(27) isolados com resistência a oxacilina de 48,4%(28) e isolados com resistência intermediária à vancomicina 9,75%.(27) Já S. aureus, no estudo realizado por Souza,(17) apresentou resistência de 80% a oxacilina, não apresentando resistência à vancomicina. Staphylococcus coagulase negativa também apresentou resistência aos outros grupos de antibióticos como: quinolonas (48%), sulfonamidas (34%) e em menor intensidade aos aminoglicosídeos (2%). Já S. aureus apresentou 10% de resistência aos aminoglicosídeos e quinolonas.
CONCLUSÃO
Esses dados revelam presença de bactérias resistentes e multirresistentes entre os isolados identificados. Diante do exposto, mostra a importância da realização de trabalhos que identifiquem o perfil de prevalência de microrganismos, resistentes e multirresistentes. Essas informações são importantes para o planejamento de controle de infecção hospitalar para o Comissão de Controle de Infecção Hospitalar. Assim contribuindo com a diminuição de morbidade, mortalidade e o alto custo financeiro diante dos internamentos prolongados dos pacientes em ambientes hospitalares devido aos quadros de sepse.
Agradecimento
Os autores agradecem à Universidade de Pernambuco, ao Pronto Socorro Cardiológico de Pernambuco e ao Laboratório de Análises Clínicas do Procape pelo apoio e colaboração para a construção deste trabalho.
Abstract
Objective: Sepsis is involved with the main causes of morbidity and mortality in hospitalized patients. These patients are vulnerable to this type of infection due to various factors such as length of stay, invasive procedures, recurrent infections, and antibiotic therapy prolonged. Therefore, this study aimed to identify the microbiological and resistance profile in positive blood cultures of patients admitted to the Cardiac Emergency of Pernambuco (Procape) in 2017. Methods: Blood cultures from January to December 2017 were analyzed. Blood cultures were processed on BactT/Alert® 3D automation equipment, microbial detection systems and then identified by Biomerieux® Vitek 2 compact. Results: From a total of 3,323 blood culture samples sent to the Hospital Laboratory, the prevalence of positivity of 120 (3.62%) samples was verified, of which there was the prevalence of K. pneumoniae 18 (15%), followed for S. haemolyticus 17 (14.16%), shortly after, S. epidermidis 16 (13.33%). Several bacteria showed multiresistance profile such as E. cloacae, A. baumannii, K. pneumoniae, P. aeruginosa, S. aureus and coagulase negative Staphylococcus. Conclusion: Results demonstrated the presence of resistant and multiresistant bacteria, with different resistance profiles. It is important to know bacterial resistance profile, aiming at the adequate treatment of patients with sepsis, preventing hospital infections.
Keywords
Bacteria; drug resistance; sepsis; hemoculture
REFERÊNCIAS
- Hotchkiss RS, Karl IE. The pathophysiology and treatment of sepsis. N Engl J Med. 2003 Jan 9;348(2):138-50. doi: 10.1056/NEJMra021333.
- Siqueira-Batista R, Gomes AP, Calixto-Lima L, Vitorino RR, Perez MCA, Mendonça EG de, et al. Sepse: atualidades e perspectivas. Rev. bras. ter. intensiva [Internet]. 2011 June ; 23(2): 207-216. Available from: http://www.scielo.br/scielo.php?script=sci_ arttext&pid=S0103-507X2011000200014&lng=en. https://doi.org/10.1590/S0103-507X2011000200014.
- Silva E, Pedro Mde A, Sogayar AC, Mohovic T, Silva CL, Janiszewski M, et al; Brazilian Sepsis Epidemiological Study. Brazilian sepsis epidemiological study (BASES study). Crit Care. 2004 Aug;8(4):R251-60. doi: 10.1186/cc2892.
- Degoricija V, Sefer S, Kujundzic-Tiljak M, Gjurasin M. Intensive care units in Croatia: 2001 survey. Croat Med J. 2002 Dec;43 (6):713-21.
- Sousa MA, et al. Hemoculturas positivas de pacientes da Unidade de Terapia Intensiva de um hospital escola de Goiânia-GO, entre 2010 e 2013. Revista EVS – Revista de Ciências Ambientais e Saúde, v. 41, n. 3, p. 627-635, 2014.
- Aguiar E. História natural da sepse:[revisão]. Brasília méd, v. 47, n. 1, 2010.
- Fernandes AP, Silva CJ, Costa C, Schreiber AZ, Mello FA, Teixeira-Loyola ABA. Incidência Bacteriana em Hemoculturas no Hospital das Clínicas Samuel Libânio de Pouso Alegre MG. Revista Eletrônica Acervo Saúde/Electronic Journal Collection Health v. 3, p. 122-133, 2011.
- Taniguchi LU, Azevedo LCP, Bozza FA, Cavalcanti AB, Elaine Maria Ferreira EM, Carrara FSA, et al. Availability of resources to treat sepsis in Brazil: a random sample of Brazilian institutions. Rev Bras Ter Intensiva. 2019 May 30;31(2):193-201. doi: 10.5935/0103-507X.20190033. [Article in Portuguese, English]
- Lobo SM, Rezende E, Mendes CL, Oliveira MC. Mortality due to sepsis in Brazil in a real scenario: the Brazilian ICUs project. Rev Bras Ter Intensiva. 2019;31(1):1-4. doi: 10.5935/0103-507X.20190008. [Article in Portuguese, English].
- Guilarde AO, Turchi MD, Martelli CMT. Bacteremias em pacientes internados em hospital universitário. Rev Assoc Med Bras, v. 53, n. 1, p. 34-8, 2007. https://doi.org/10.1590/S0104-42302007000100016.
- Barros LM, Bento JNC, Caetano JA, Moreira RAN, Pereira FGF, Frota NM, et al. Prevalência de micro-organismo e sensibilidade antimicrobiana de infecções hospitalares em unidade de terapia intensiva de hospital público no Brasil. Revista de Ciências Farmacêuticas Básica e Aplicada, v. 33, n. 3, p. 429-435, 2012.
- Araujo MRE. Hemocultura: recomendações de coleta, processamento e interpretação dos resultados. J Infect Control 2012; 1 (1): 08-19.
- Costa ALP, Junior ACSS. Resistência bacteriana aos antibióticos e Saúde Pública: uma breve revisão de literatura. Estação Científica (UNIFAP), v. 7, n. 2, p. 45-57, 2017.
- Meneghetti BH, Salla A, Oliveira LT, Righi R, Alves SH. Epidemiologia das infecções bacterianas e fúngicas diagnosticada através de hemocultivos, no Hospital Universitário de Santa Maria-HUSM, RS. Rev. bras. anal. clin. 2004;36(3):173-175.
- Ruschel DB, Rodrigues AD, Formolo F. Perfil de resultados de hemoculturas positivas e fatores associados. Rev. bras. anal. cli. 2017;49(2): 158-63.
- Santana TR, Lima Jr AA, Lobo IMF, Araújo JG. Infecção de corrente sanguínea em um hospital terciário. Rev Soc Bras Clin Med, v. 14, n. 1, p. 22-6, 2016.
- Sousa NA, Coelho CGV, Mesquita CHS, Pires FGB, Rosa PB, Brito ILP. Sepse neonatal – perfil microbiológico e sensibilidade antimicrobiana em um hospital no Nordeste do Brasil. Rev. bras. anal. clin., [s.l.], v. 51, n. 1, p.46-51, 2019.
- Nordmann P, Cuzon G, Naas T. The real threat of Klebsiella pneumoniae carbapenemase-producing bacteria. Lancet Infect Dis. 2009 Apr;9(4):228-36. doi: 10.1016/S1473-3099(09)70054-4
- Nguyen M, Eschenauer GA, Bryan M, O’Neil K, Furuya EY, Della-Latta P, et al. Carbapenem-resistant Klebsiella pneumoniae bacteremia: factors correlated with clinical and microbiologic outcomes. Diagn Microbiol Infect Dis. 2010 Jun;67(2):180-4. doi: 10.1016/j.diagmicrobio.2010.02.001.
- Carissa D, et al. Extended-spectrum b-Lactamase producing Escherichia coli strain of poultry origin in Owerri, Nigeria. World J Med Sci, v. 8, n. 4, p. 349-354, 2013.
- Rasheed JK, Tenover FC. Detection and characterization of antimicrobial resistance genes in pathogenic bacteria. In: Manual of Clinical Microbiology, 10th Edition. American Society of Microbiology, 2011. p. 1239-1261.
- Moura Pinto F, Simas DM, Baldin CP, Limberger II, Silva RCF, Antochevis LC, Vieira FJ, et al. Prevalência de carbapenemases em enterobactérias resistentes a carbapenêmicos em quatro hospitais terciários de Porto Alegre [Prevalence of carbapenemases in carbapenem-resistant Enterobacteriaceae in four tertiary care hospitals in Porto Alegre]. Clinical & Biomedical Research, v. 34, n. 1, 2014.
- Alves AP, Behar PRP. Infecções hospitalares por enterobactérias produtoras de KPC em um hospital terciário do sul do Brasil. Rev. AMRIGS, v. 57, n. 3, p. 213-218, 2013.
- Queiroz GM, Silva LM, Pietro RCLR, Salgado HRN. Multirresistência microbiana e opções terapêuticas disponíveis. Rev Bras Clin Med. São Paulo, 2012 mar-abr;10(2):132-8
- Raja NS, Singh NN. Antimicrobial susceptibility pattern of clinical isolates of Pseudomonas aeruginosa in a tertiary care hospital. J Microbiol Immunol Infect. 2007 Feb;40(1):45-9.
- Pires EJ, Silva Júnior VV, Lopes AC, Veras DL, Leite LE, Maciel MA. Epidemiologic analysis of clinical isolates of Pseudomonas aeruginosa from an university hospital. Rev Bras Ter Intensiva. 2009 Dec;21(4):384-90. [Article in English, Portuguese]
- Melo GB, Melo MC, Carvalho KS, Gontijo Filho PP. Staphylococcus aureus e estafilococos coagulase negativos resistentes à vancomicina em um Hospital Universitário Brasileiro. Rev Ciênc Farm Básica Apl.,2009;30(1): 45-50.
- Rigatti F, Tizotti MK, Hörner R, Domingues VO, Martini R, Mayer LE, et al. Bacteremias por Staphylococcus coagulase negativos oxacilina resistentes em um hospital escola na cidade de Santa Maria, Estado do Rio Grande do Sul. Rev. Soc. Bras. Med. Trop. [online]. 2010, vol.43, n.6, pp.686-690. https://doi.org/10.1590/S0037-86822010000600017.
Correspondência
Vera Lúcia Nascimento Bezerra
Universidade de Pernambuco
Pronto Socorro Cardiológico (Procape)
- dos Palmares, s/n – Santo Amaro
74970-240 – Recife-PE, Brasil
