Principais variantes do SARS-CoV-2 notificadas no Brasil
Main SARS-CoV-2 variants notified in Brazil
Cleonice Maria Michelon1
1Doutora, Departamento de Análises Clínicas – Universidade Federal de Santa Catarina. Florianópolis-SC, Brasil.
Recebido em 10/03/2021
Aprovado em 29/04/2021
DOI: 10.21877/2448-3877.202100961
INTRODUÇÃO
O SARS-CoV-2 (do inglês Severe Acute Respiratory Syndrome CoronaVirus-2), agente etiológico da COVID-19 (do inglês COronaVIrus Disease 2019), pertence à família dos Coronavírus, que são um grupo de vírus envelopados que possuem RNA linear de fita simples sentido positivo. O SARS-CoV-2 apresenta quatro proteínas estruturais principais: a glicoproteína de superfície (do inglês spike) (S), a proteína do envelope (E), a proteína da matriz (M) e a proteína do nucleocapsídeo (N).(1)
Desde o início da pandemia de COVID-19, análises genéticas do SARS-CoV-2 em vários países e em diferentes momentos revelaram que o vírus sofreu diversas mutações.(2–5) As mutações são eventos naturais da replicação viral, sendo muito mais frequentes em vírus de genoma RNA. A maior parte das mutações é neutra, ou seja, não fornece qualquer vantagem ou desvantagem para o vírus. Entretanto, um pequeno número de mutações pode conferir novas propriedades químicas às proteínas virais, resultando em mudanças na forma como o vírus se comporta nas infecções.(6)
As mutações na proteína S apresentadas pelo SARS-CoV-2 são as mais relevantes do ponto de vista clínico-epidemiológico, considerando seu importante papel no processo infeccioso.(7) A proteína S é uma grande glicoproteína transmembrana presente na superfície externa dos Coronavírus. A proteína é clivada por proteólise formando as subunidades S1, responsável pela adsorção do vírus à superfície da célula, e S2, que promove a fusão do envelope do vírus com a membrana plasmática da célula.(8) A infecção celular acontece pela interação do domínio RDB (do inglês receptor-binding domain) da subunidade S1 do vírus com o receptor da enzima conversora de angiotensina-2 (ECA-2) da célula alvo.(7,8)
Mutações na proteína S que aumentam a afinidade do vírus pelo receptor celular ou a quantidade de vírus eliminada durante o curso da infecção podem estar associadas ao aumento da transmissão do vírus.(7,9–11) Exemplos são as mutações D614G, associadas a elevada carga viral e maior transmissibilidade,(11) e 501Y, também associada a maior transmissibilidade.(8,10,12)
O acesso público global aos bancos de dados e plataformas genômicas, como Global Initiative on Sharing All Influenza Data (GISAID), GenBank, Genome Warehouse (NGDC) e National Microbiology Data Center (NMDC), permitiu o rastreamento em tempo real da emergência e disseminação das mutações identificadas no SARS-CoV-2 nas diferentes partes do mundo. Nesse contexto, a presente revisão teve como objetivo descrever as principais variantes do SARS-CoV-2 notificadas no Brasil. Para tanto, foi realizada uma busca na literatura sobre a identificação de variantes de preocupação e de interesse em nosso país, com foco em suas principais características e seus possíveis impactos sobre a saúde humana.
CARACTERIZAÇÃO GENÔMICA DO SARS-CoV-2
O sequenciamento genômico do SARS-CoV-2 e a divulgação oportuna dos dados levaram à caracterização do agente etiológico envolvido no surto inicial, bem como ao desenvolvimento de protocolos diagnósticos e ao acompanhamento da evolução da pandemia de COVID-19. O sequenciamento genômico tornou-se uma ferramenta essencial para gerar dados virológicos de SARS-CoV-2, permitindo a detecção das diversas mutações.(13)
Apesar das denominações clado, linhagem e variante não corresponderem a uma hierarquia taxonômica oficial, desde sua caracterização genômica, o SARS-CoV-2 foi classificado em diferentes grupos genéticos ou clados e subdividido em categorias de acordo com o sistema de nomenclatura utilizado. Mutações específicas definem os grupos genéticos virais (também denominados linhagens), enquanto o surgimento de mutações adicionais, promovendo diferenças dentro de cada grupo genético, dá origem às denominadas variantes.(14) Para facilitar a compreensão e o acompanhamento das evoluções genéticas do SARS-CoV-2 e reduzir estigmas geográficos, a Organização Mundial da Saúde (OMS) criou um novo sistema de nomenclatura unificado, que usa o alfabeto grego, para classificar as variações do vírus.(15)
A partir da identificação do SARS-CoV-2 até março de 2021, mais de 845 mil sequências genômicas completas foram compartilhadas através de bases de dados de acesso público.(16) Com base nesses dados foram identificadas diversificações genéticas agrupadas em três diferentes clados. Dentro de cada clado, diferentes linhagens e variantes foram observadas, destacando uma crescente modificação do vírus SARS-CoV-2 ao longo do tempo. A cocirculação de diferentes clados genéticos foi observada em vários países, bem como a migração de linhagens genéticas da Europa para a América do Sul.(17) Entretanto, é importante salientar que as informações sobre os clados predominantes em nível global devem ser interpretadas com cautela, devido a representação enviesada dos dados, com maior participação de países de alta renda.(16)
O grupo de trabalho da OMS que acompanha a evolução do SARS-CoV-2, avaliando diversos fatores incluindo transmissibilidade, virulência, alterações fenotípicas e propagação, classificou as variantes circulantes globalmente em variantes de preocupação (VOC, do inglês, variant of concern) e variantes de interesse em saúde pública (VOI, do inglês variant of interest). Foram classificadas no grupo das VOC as variantes Alfa (B.1.1.7), identificada inicialmente no Reino Unido, Beta (B.1.351), descoberta na África do Sul, Gama (B.1.1.28.1), originária do Brasil (Manaus) e Delta (B.1.617.2), identificada na Índia. No grupo das VOI as variantes Eta (B.1.525), detectada em diversos países, Epsilon (B.1.427/B.1.429), identificada nos Estados Unidos da América (Califórnia), Zeta (B.1.1.28.2), originária do Brasil (Rio de Janeiro), Teta (B.1.1.28.3), detectada nas Filipinas e no Japão, Iota (B.1.526), descoberta nos Estados Unidos da América, Kapa (B.1.617.1) detectada na Índia, e Lambda (C.37), originária do Peru(18) (Quadro 1).
Quadro 1 – Variantes de interesse e variantes de preocupação do SARS-CoV-2.
| OMS | Nextrain
clado |
Pango
linhagem |
GISAID
clado |
Primeira detecção | Primeiras amostras | Mutações características |
| Variantes de preocupação (VOC) | ||||||
| Alfa
|
20J/501Y.V1 | B.1.1.7 | GRY GR/501Y.V1 | Reino Unido | Set 2020 | H69/V70del, Y144del, N501Y, A570D, P681H, S106/G107/F108del |
| Beta | 20H/501Y.V2 | B.1.351 | GH/501Y.V2 | África do Sul | Ago 2020 | L242/A243/L244del, K417N, E484K, N501Y, S106/G107/F108del |
| Gama | 20J/501Y.V3 | B.1.1.28.1
conhecida como P.1 |
GR/501Y.V3 | Brasil e Japão | Dez 2020 | K417T, E484K, N501Y, S106/G107/F108del |
| Delta | 21A/S:478K | B.1.617.2 | G/452R.V3 | Índia | Out 2020 | T19R, (G142D), 156del, 157del, R158G, L452R, T478K, D614G,
P681R, D950N |
| Variantes de interesse (VOI) | ||||||
| Epsilon | 20C/S.452R | B.1.427/ B.1.429 | GH/452R.V1 | Estados Unidos da América | Jun 2020 | L452R, W152C, S13I, D614G |
| Zeta | 20B/S.484K | B.1.1.28.2
conhecida como P.2 |
GR | Brasil | Abril 2020 | L18F, T20N, P.26S, F157L, E484K, D614G, S929I, V1176F |
| Eta | 20A/S484K | B.1.525 | G/484K.V3 | Múltiplos países | Dez 2020 | H69-V70del, Y144del, Q52R, E484K, Q677H, D616G, S929I, V1176F |
| Teta | 20B/S:265C | B.1.1.28.3
conhecida como P.3 |
GR | Filipinas e Japão | Fev 2021 | 141-143del, E484K, N501Y, P681H |
| Iota | 20C/S:484K | B.1.526 | GH | Estados Unidos da América | Nov 2020 | LSF, T95I, D253G, D614G, V483A, H655Y, G669S, Q949R, N1187D |
| Kapa | 21A/S:154K | B.1.617.1 | G/452R.V3 | Índia | Out 2020 | (T95I), G142D, E154K, L452R, E484Q, D614G, P681R, Q1071H |
| Lambda | 21G | C.37 | GR/452Q.V1 | Peru | Dez 2020 | 246-252del, G75V, T76I, L452Q, F490S, D614G e T859N |
Fonte: Adaptado de COVID-19 Weekly Epidemiological Update. WHO, 13 july 2021. (https://www.who.int/publications/m/item/weekly-epidemiological-update-on-covid-19—13-july-2021). Consultado em 14/07/2021.
LINHAGENS E VARIANTES CIRCULANTES NO BRASIL
No Brasil, no período de janeiro de 2020 a maio de 2021 foram registradas mudanças na frequência das linhagens dominantes, segundo dados da Rede Genômica Fiocruz (Figura 1). Inicialmente a epidemia foi impulsionada principalmente pelas linhagens B.1.1.28 e B.1.1.33, que foram as mais prevalentes até outubro de 2020.(19) Após esse período, destaca-se a circulação de duas variantes de origem nacional, P.1 e P.2, originadas da linhagem B.1.1.28.(16) As duas variantes brasileiras juntas, em apenas quatro meses após sua emergência, corresponderam a 75% dos sequenciamentos no território nacional.(7) No que tange a notificação das variantes de preocupação e de interesse em saúde pública, foram registradas no Brasil, até julho de 2021, as quatro variantes classificadas como VOC e duas (Zeta e Lambda) das sete variantes classificadas como VOI pela OMS.
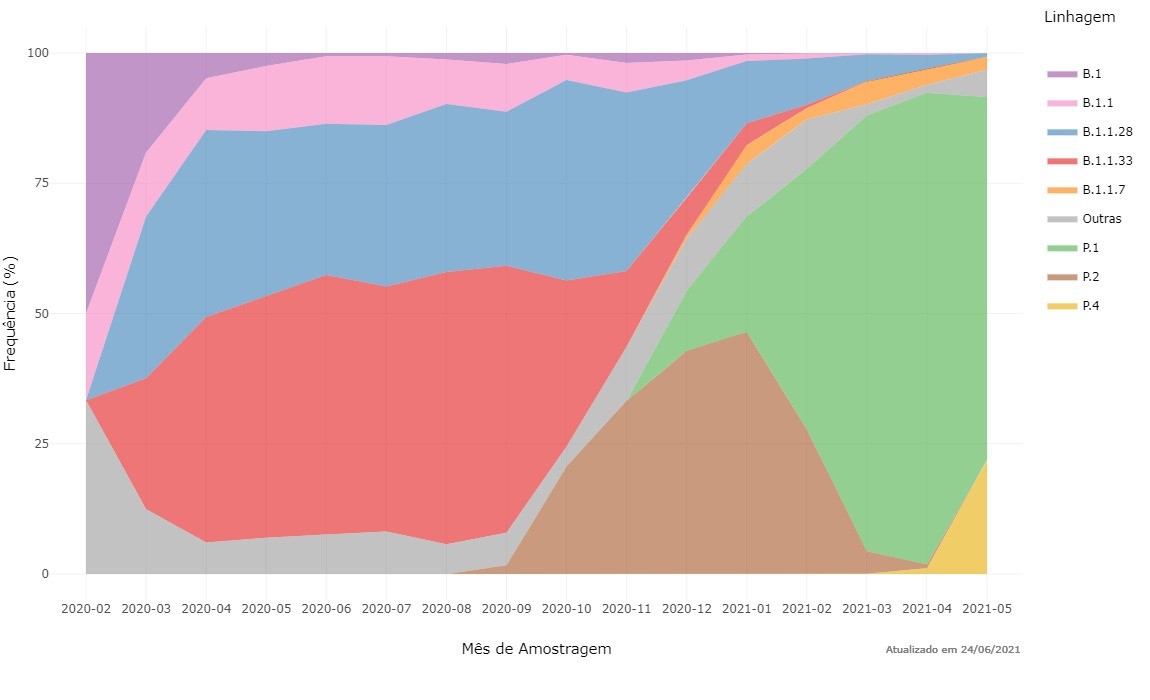
Figura 1. Frequência das principais linhagens de SARS-CoV-2 por mês de amostragem (janeiro/2020 a maio/2021) no Brasil.
Fonte: Rede Genômica – Fiocruz. (http://www.genomahcov.fiocruz.br/frequencia-das-principais-linhagens-do-sars-cov-2-por-mes-de-amostragem/). Consultado em 14/07/2021.
VARIANTES DE PREOCUPAÇÃO E DE INTERESSE REGISTRADAS NO BRASIL
ALFA (Variante B.1.1.7 – Variante do Reino Unido)
A variante Alfa (também denominada B.1.1.7, 20B/501Y.V1 ou VOC-202012/01) foi detectada em setembro de 2020, tornando-se a linhagem dominante no Reino Unido em dezembro de 2020, mesmo mês em que foi identificada no Brasil. Essa variante apresenta sete mutações na proteína S, incluindo a mutação N501Y que está associada a maior afinidade do vírus pelo receptor ECA-2,(8,10,12) o que pode explicar a sua rápida expansão,(10,12) bem como a maior resistência à neutralização por anticorpos.(20) A mutação N501Y caracteriza-se pela substituição do aminoácido asparagina pela tirosina na posição 501, no domínio de ligação ao receptor (RDB) da proteína S.(10) Essa mutação aumentou em sete vezes a afinidade do vírus pelo receptor ECA-2.(8) Um estudo realizado por Supasa e colaboradores que testou a capacidade neutralizante do soro obtido de pacientes convalescentes de infecção pelo SARS-CoV-2, linhagem Wuhan, e soro de indivíduos que receberam vacina da Oxford-AstraZeneca e Pfizer contra o vírus B.1.1.7 concluiu que a variante exigia maiores concentrações de soro para atingir a neutralização. Entretanto, o escape generalizado de anticorpos monoclonais e anticorpos gerados por infecção natural ou vacinação não foi observado.(20)
Além da mutação N501Y, essa variante carrega algumas deleções que resultaram na perda de aminoácidos no domínio N-terminal (NTD) da proteína S. Essas deleções também podem contribuir para o escape da resposta imune,(20) considerando que o NTD constitui um super sítio de ligação de anticorpos neutralizantes.(21) A perda desses aminoácidos pode influenciar também na sensibilidade de kits diagnósticos que empregam sondas voltadas à detecção desta sequência. Devido à deleção, sondas usadas em ensaios comerciais perdem a capacidade de se ligar ao amplicon, gerando resultado falso negativo.(22)
A variante foi considerada mais transmissível(10,23) e mais letal(24,25) quando comparada as cepas dominantes registradas no Reino Unido anteriormente. Um estudo de coorte encontrou risco relativo de morte de 1,64 (IC de 95% 1,32 a 2,04) entre indivíduos infectados com o vírus B.1.1.7 comparado com outras linhagens.(24) Estudos observacionais retrospectivos também estimaram um risco aumentado de morte (35%) associado à variante.(25) Não existem até o momento evidências de doença mais grave em crianças e jovens acometidos pela variante Alfa,(26) apesar dos registros de aumento na proporção de casos com necessidade de hospitalização em indivíduos com menos de 60 anos.(27)
BETA (Variante B.1.351 – Variante Sul-Africana)
A variante Beta (B.1.351 ou 501Y.V2) surgiu na África do Sul, no início de outubro de 2020 e se espalhou rapidamente para regiões próximas. A variante foi identificada após a primeira onda da epidemia em uma área metropolitana gravemente afetada, Nelson Mandela Bay, localizada na costa da Província do Cabo Oriental, de onde se espalhou tornando-se dominante, em poucas semanas, nas províncias de Eastern Cape e Western Cape e na Europa Ocidental.(9) Até fevereiro de 2021 a variante já era detectada em 35 países,(28) entretanto, a confirmação de registros dessa variante no Brasil ocorreu somente no final de abril do mesmo ano.(29)
Comparada à cepa de referência do vírus SARS-CoV-2 (cepa de Wuhan), a variante apresenta 12 mutações não sinônimas e uma deleção. A maioria das mutações (7) e deleção (1) encontradas está localizada na proteína S, enquanto as demais estão localizadas nas regiões ORF1a, proteína E e proteína N.(28) O alto número de mutações nas regiões imunodominantes da proteína S, incluindo três (K417N, E484K e N501Y) no RBD(9,28) e quatro na região NTD, sugere possibilidade de escape dos anticorpos neutralizantes.(28)
A mutação N501Y, conforme descrito anteriormente, está relacionada a maior afinidade do vírus pelo receptor ECA-2(8,10,12) e a maior resistência à neutralização por anticorpos.(20) As mutações E484K e K417N, presentes também no RBD da proteína S, desempenham papel fundamental na interação do vírus com o receptor e na evasão imunológica.(28) Na mutação E484K, o ácido glutâmico (aminoácido com carga elétrica negativa) é substituído por uma lisina (aminoácido carregado positivamente). Essa mudança de carga na região flexível do RBD parece criar uma interação significativamente mais forte com o receptor celular.(30) Por sua vez, a mutação K417N (substituição da lisina por asparagina) parece influenciar minimamente a interação do vírus com a célula alvo.(28) Entretanto, o conjunto das três mutações intensifica a afinidade do vírus pelo receptor, aumentando a transmissibilidade e permitindo escape da neutralização por anticorpos anti-SARS-CoV-2.(30) As demais mutações presentes na proteína S da variante se encontram na região NTD, que também é uma região alvo de anticorpos neutralizantes. Estudos destacam que a combinação de mutações nas regiões RBD e NTD da proteína S afeta significativamente a neutralização da variante Beta por anticorpos monoclonais e soros imunes obtidos de pacientes convalescentes ou vacinados.(8,31–33)
Em relação à gravidade da doença produzida pela variante Beta, apesar de um estudo realizado na Europa ter demonstrado que os pacientes infectados com a variante tiveram maior necessidade de admissão em unidades de terapia intensiva, quando comparados aos infectados pelas outras VOC,(27) não existem, até o momento, evidências robustas que comprovem maior grau de virulência ou maior gravidade da doença relacionada a variante Beta.(34)
GAMA (Variante B.1.1.28.1 ou P.1 – Variante Brasileira – Manaus)
Essa variante foi notificada à OMS em janeiro de 2021 pelo Japão, após detecção em quatro viajantes provenientes do Brasil.(7,14) A variante Gama (também denominada linhagem P.1) identificada em dezembro de 2020 em Manaus, capital do estado do Amazonas, esteve ausente nas amostras coletadas até novembro de 2020. Entretanto, em dezembro do mesmo ano, a variante foi identificada em 42% das amostras e em 91% em janeiro de 2021 no mesmo município, sugerindo transmissão local.(7,35) Até 20 de abril de 2021, a variante Gama já havia sido registrada em 43 países, segundo boletim epidemiológico semanal publicado pela OMS.(15)
A P.1 apresenta uma constelação única de mutações, incluindo alterações genéticas importantes do ponto de vista clínico-epidemiológico.(35) A P.1 compartilha a mutação N501Y, relacionada a maior transmissão, com as variantes Alfa e Beta, além de exibir mais duas mutações no RBD da proteína S, a mutação E484K e a mutação K417T. As mutações citadas estão associadas à evasão da resposta imune.(8,9,35,36) Wang e colaboradores identificaram que a linhagem P.1 é refratária a diversos anticorpos monoclonais, apresentando também maior resistência à neutralização por plasma convalescente e soros obtidos de indivíduos vacinados.(36) Segundo alguns autores, essa nova linhagem está relacionada a maior transmissibilidade e maior risco de reinfecção por Coronavírus.(35,36)
Um estudo que comparou dados de pacientes de sete países europeus infectados por VOC e não-VOC identificou maior necessidade de internação hospitalar em pacientes com a variante P.1 (20%) quando comparados aos infectados por outras variantes.(27) Outro estudo demonstrou, ainda, que simultaneamente ao surgimento da linhagem P.1 no estado do Amazonas houve aumento na mortalidade de indivíduos com COVID-19 na faixa etária dos 20 aos 50 anos, em ambos os sexos. Esses dados sugerem que as mutações presentes na variante P.1 podem estar associadas a alterações na virulência e na patogenicidade do vírus.(37)
ZETA (Variante B.1.1.28.2 ou P.2 – Variante Brasileira – Rio de Janeiro)
Essa variante, também denominada linhagem P.2, foi identificada em outubro de 2020 no estado do Rio de Janeiro. Após sua detecção, o sequenciamento de genomas coletados de vários municípios do estado, entre abril e novembro, detectou a presença da variante em amostras do mês de julho de 2020, cerca de quatro meses antes de sua identificação.(38) A proporção dessa variante aumentou significativamente entre setembro de 2020, quando correspondia a 0,7% das amostras, e fevereiro de 2021, quando foi detectada em 45% das amostras sequenciadas no país.(14) Em apenas quatro meses a P.2 foi notificada em 16 países.(38) Nas Américas, a variante foi identificada na Argentina, no Chile, nos Estados Unidos da América, no México, na ilha de São Martinho, no Uruguai e na Venezuela.(16)
A P.2 é caracterizada por cinco mutações definidoras e outras cinco mutações presentes na maioria dos genomas.(38) A P.2 apresenta somente uma mutação na proteína S, a mutação E484K no RBD. Essa mutação está associada ao aumento da interação do vírus com o receptor ECA-2 e ao escape de anticorpos neutralizantes contra o SARS-CoV-2.(7,38,39)
Dentre as mutações presentes no RBD, a mais preocupante parece ser a E484K. E484 é o local onde as mutações tendem a ter o maior efeito sobre a ligação de anticorpos.(40) Estudos mostraram que as mutações no local E484 reduzem a potência de neutralização de alguns plasmas humanos em mais de 10 vezes, embora outros plasmas não sejam afetados por mutações neste local.(40,41) A resistência à neutralização das variantes que apresentam a mutação E484K por anticorpos pode explicar os recentes casos de reinfecção pela P.2 observados no Brasil.(42,43)
DELTA/KAPA (Variante B.1.617 – Variante Indiana)
A variante B.1.617 foi identificada primeiramente na Índia em dezembro de 2020, embora uma versão anterior tenha sido detectada em outubro de 2020. Em abril de 2021 a variante já havia sido notificada em 21 países.(44) Segundo os últimos dados da OMS, a variante foi classificada em três linhagens, denominadas B.1.617.1, B.1.617.2 e B.1.617.3, com pequenas diferenças e distinta distribuição geográfica.(18)
No Brasil, os primeiros casos de COVID-19 causados pela variante indiana foram confirmados na segunda quinzena de maio de 2021. Inicialmente, um caso no estado do Maranhão, seguido por casos notificados no Rio de Janeiro, em Minas Gerais, no Paraná e em Goiás,(45) totalizando mais de 100 casos até meados de julho de 2021.
Na variante B.1.617 foram identificadas 13 mutações que resultaram em substituições de aminoácidos.(44) As mutações que mais preocupam são as encontradas na proteína S, incluindo duas mutações na região NTD (G142D e E154K), duas na região RBD (L452R e E484Q) e uma no local de clivagem da furina polibásica no limite S1/S2 (P681R).(46)
As mutações na região RBD combinadas com a mutação no local de clivagem da furina podem resultar em aumento na ligação do vírus à ECA-2 e maior taxa de clivagem S1/S2, resultando em maior transmissibilidade.(44) Além disso, um estudo demonstrou que apesar da maioria dos soros de indivíduos convalescentes e vacinados com Pfizer e Moderna ter capacidade de neutralizar a variante B.1.617, esta se mostrou seis a oito vezes mais resistente à neutralização quando comparada à cepa WA1/2020, cepa de referência para estudo do SARS-CoV-2 nos Estados Unidos da América.(46) Outro estudo demonstrou que soros de indivíduos convalescentes e soros de indivíduos imunizados com as vacinas Pfizer-BioNTech e Oxford-AstraZeneca mostraram neutralização reduzida das variantes B.1.617.2 (Delta) e B.1.617.1 (Kapa). O mesmo estudo sugeriu ainda que indivíduos infectados anteriormente com as variantes B.1.351 (Beta) e P.1 (Gama) são provavelmente mais suscetíveis à reinfecção pela cepa Delta.(47)
LAMBDA (Variante C.37 – Variante Andina)
A variante Lambda foi relatada pela primeira vez em Lima, capital do Peru, em dezembro de 2020, quando correspondia a 0,5% dos genomas avaliados, expandindo para 96,6% em abril de 2021.(48) Em meados de junho de 2021, já era encontrada em 26 países nas Américas, na Europa e na Oceania. Esta variante, que tem elevada prevalência em alguns países da América do Sul, foi identificada no Brasil em fevereiro de 2021, no estado de São Paulo, e desde então tem sido apenas ocasionalmente notificada.(49)
A variante Lambda apresenta a deleção 3575-3677 no gene ORF1a, também presente nas variantes Alfa, Beta e Gama. Além disso, a variante apresenta uma deleção (246-252) e múltiplas mutações não sinônimas (G75V, T76I, L452Q, F490S, D614G e T859N) no gene da proteína S. As mutações L452Q e F490S estão na região RBD da proteína S.(49)
A mutação L452Q é similar à L452R, presente nas variantes Delta, Epsilon e Kapa, associada ao escape da neutralização por anticorpos e maior infectividade devido ao aumento da afinidade para o receptor ECA-2. A mutação F490S também foi associada ao escape da neutralização por anticorpos.(48) Um estudo in vitro realizado no Chile demonstrou neutralização por anticorpos 3,05 vezes menor para a variante Lambda quando comparada à linhagem selvagem. O estudo utilizou plasma de doadores que receberam duas doses da vacina Coronavac. O mesmo estudo demonstrou, ainda, aumento na infecciosidade da variante Lambda, quando comparada às variantes Alfa e Gama.(50)
OUTRAS VARIANTES RECENTEMENTE IDENTIFICADAS NO BRASIL
Variantes da linhagem B.1.1.33
A linhagem B.1.1.33 foi uma das principais e mais difundidas no Brasil no ano de 2020. Diversas variantes dessa linhagem já foram definidas, entretanto nenhuma delas apresentava mutação preocupante na proteína S.(51) Recentemente foram identificadas no Brasil duas novas variantes, cujas inferências evolutivas apontam para a ancestral B.1.1.33: a N9, que apresenta a ocorrência da mutação E484K,(51,52) e a N10, que apresenta 14 mutações definidoras de linhagem, incluindo mutações no RBD (E484K e V445A) e três deleções no NTD da proteína S.(53)
Variantes da linhagem B.1.1.28
Além das variantes P.1 e P.2, estudos apontam duas possíveis novas variantes, uma denominada NP.13L e a outra denominada P.4, que também descendem da linhagem B.1.1.28, uma das linhagens predominantes no Brasil. Descrita pela primeira vez em janeiro de 2021, no sul do Brasil, a variante NP.13L foi detectada também na Paraíba e na Bahia, além da Inglaterra, do Japão e da Holanda.(52) A NP.13L é caracterizada por 12 mutações definidoras de linhagem, incluindo a mutação E484K na proteína S.(54) Já a variante P.4 foi identificada em maio de 2021 no interior do estado de São Paulo. Essa variante, reconhecida pela Sociedade Brasileira de Virologia, apresenta a mutação L452R na proteína S, mutação também presente na variante indiana.(55)
CONCLUSÃO
Desde o surgimento dos primeiros casos de infecção pelo SARS-CoV-2 até o presente momento, estamos sendo constantemente confrontados com o surgimento de novas variantes ao redor do mundo. No Brasil, foram notificados casos de COVID-19 por diversas linhagens, incluindo as quatro variantes de preocupação e duas variantes de interesse, segundo classificação da OMS. Importante destacar que, destas, duas são de origem nacional (P.1 e P.2) e novas variantes estão sendo caracterizadas.
As principais características das novas variantes consistem em maior afinidade de ligação ao receptor celular, característica que está relacionada a maior transmissão, presente em todas as variantes de preocupação; resistência aumentada a anticorpos neutralizantes, característica mais significativa nas variantes Beta (B.1.351), Gama (P.1) e Delta (B.1.617.2); aumento da virulência, observado principalmente na variante Alfa (B.1.1.7); risco aumentado de reinfecção, principalmente para as variantes Gama (P.1) Zeta (P.2) e Delta (B.1.617.2).
Nesse contexto, a manutenção de medidas preventivas como o uso de máscaras, distanciamento social e ampliação da vacinação, para minimizar a circulação do vírus, prevenindo infecções e consequentemente reduzindo as possibilidades de evolução do SARS-CoV-2, é imperativa para o controle da pandemia. O monitoramento genético constante das cepas de SARS-CoV-2 também se faz necessário, pois além de permitir a rápida identificação do surgimento de novas variantes, contribui para o entendimento da dinâmica da pandemia e para a produção de novas vacinas e testes diagnósticos.
Abstract
The emergence of SARS-CoV-2 (Severe Acute Respiratory Syndrome Coronavirus 2) variants in a lot of countries raises concern for health authorities around the world. The SARS-CoV-2 variants present mutations, mainly in the S protein, which are related to increase transmissibility, becoming dominant in certain regions in a short time. These mutations also seem to be associated with higher virulence, resistance to both monoclonal antibodies and those produced in response to previous infection or vaccine and to the most frequency of re-infections. In this review, the Alpha, Beta, Gamma, Delta, Zeta and Lambda variants classified as concern and interest variants, according to the World Health Organization, was described. These variants have been reported in Brazil until now, and the review addresses their main characteristics and possible impacts on human health.
Keywords
SARS-CoV-2; variants; Brazil; COVID-19
REFERÊNCIAS
- Wang H, Li X, Li T, Zhang S, Wang L, Wu X, et al. The genetic sequence, origin, and diagnosis of SARS-CoV-2. Eur J Clin Microbiol Infect Dis [Internet]. 2020 Sep 24;39(9):1629–35. Disponível em: https://link.springer.com/10.1007/s10096-020-03899-4
- Forster P, Forster L, Renfrew C, Forster M. Phylogenetic network analysis of SARS-CoV-2 genomes. Proc Natl Acad Sci [Internet]. 2020 Apr 28;117(17):9241–3. Disponível em: http://www.pnas.org/lookup/doi/10.1073/pnas.2004999117
- Islam MR, Hoque MN, Rahman MS, Alam ASMRU, Akther M, Puspo JA, et al. Genome-wide analysis of SARS-CoV-2 virus strains circulating worldwide implicates heterogeneity. Sci Rep [Internet]. 2020 Dec 19;10(1):14004. Disponível em: http://www.nature.com/articles/s41598-020-70812-6
- Toyoshima Y, Nemoto K, Matsumoto S, Nakamura Y, Kiyotani K. SARS-CoV-2 genomic variations associated with mortality rate of COVID-19. J Hum Genet [Internet]. 2020 Dec 22;65(12):1075–82. Disponível em: http://www.nature.com/articles/s10038-020-0808-9
- van Dorp L, Acman M, Richard D, Shaw LP, Ford CE, Ormond L, et al. Emergence of genomic diversity and recurrent mutations in SARS-CoV-2. Infect Genet Evol [Internet]. 2020 Sep;83:104351. Disponível em: https://linkinghub.elsevier.com/retrieve/pii/S1567134820301829
- Grubaugh ND, Petrone ME, Holmes EC. We shouldn’t worry when a virus mutates during disease outbreaks. Nat Microbiol [Internet]. 2020 Apr 18;5(4):529–30. Disponível em: http://www.nature.com/articles/s41564-020-0690-4
- Freitas RRA, Giovanetti M, Alcantara LCJ. Variantes emergentes do SARS-CoV-2 e suas implicações na saúde coletiva. Interam J Med Heal [Internet]. 2021; Disponível em: http://www.ncbi.nlm.nih.gov/pubmed/33890566
- Dejnirattisai W, Zhou D, Supasa P, Liu C, Mentzer AJ, Ginn HM, et al. Antibody evasion by the P.1 strain of SARS-CoV-2. Cell [Internet]. 2021 Mar; Disponível em: https://linkinghub.elsevier.com/retrieve/pii/S0092867421004281
- Tegally H, Wilkinson E, Giovanetti M, Iranzadeh A, Fonseca V, Giandhari J, et al. Emergence and rapid spread of a new severe acute respiratory syndrome-related coronavirus 2 (SARS-CoV-2) lineage with multiple spike mutations in South Africa. 2020;2. Disponível em: https://www.medrxiv.org/content/10.1101/2020.12.21.20248640v1.full.pdf
- Leung K, Shum MH, Leung GM, Lam TT, Wu JT. Early transmissibility assessment of the N501Y mutant strains of SARS-CoV-2 in the United Kingdom, October to November 2020. Euro Surveill [Internet]. 2021;26(1). Disponível em: http://www.ncbi.nlm.nih.gov/pubmed/33413740
- Volz E, Hill V, McCrone JT, Price A, Jorgensen D, O’Toole Á, et al. Evaluating the Effects of SARS-CoV-2 Spike Mutation D614G on Transmissibility and Pathogenicity. Cell [Internet]. 2021;184(1):64-75.e11. Disponível em: http://www.ncbi.nlm.nih.gov/pubmed/33275900
- Santos J, Passos G. The high infectivity of SARS-CoV-2 B.1.1.7 is associated with increased interaction force between Spike-ACE2 caused by the viral N501Y mutation. bioRxiv Prepr [Internet]. 2021;501:1–9. Disponível em: https://www.biorxiv.org/content/10.1101/2020.12.29.424708v1
- World Health Organization – WHO, editor. Genomic sequencing of SARS-CoV-2: a guide to implementation for maximum impact on public health. Geneva; 2021. 94 p.
- Update E. Atualização epidemiológica: Variantes de SARS-CoV-2 nas Américas [Internet]. Brasília-DF: Organização Pan-Americana da Saúde – OPAS; 2021. p. 1–10. Disponível em: https://www.paho.org/
- WHO. COVID-19 Weekly Epidemiological Update 42 [Internet]. World Health Organization. 2021. p. 1-28. Disponível em: https://www.who.int/docs/default-source/coronaviruse/situation-reports/weekly_epidemiological_update_22.pdf
- Update E. Atualização epidemiológica: Variantes do SARS-CoV-2 nas Américas [Internet]. Brasília-DF: Organização Pan-Americana da Saúde – OPAS; 2021. p. 1-9. Disponível em: https://iris.paho.org/handle/10665.2/53376
- Castells M, Lopez-Tort F, Colina R, Cristina J. Evidence of increasing diversification of emerging Severe Acute Respiratory Syndrome Coronavirus 2 strains. J Med Virol. 2020;92(10):2165-72. Disponível em: https://onlinelibrary.wiley.com/doi/10.1002/jmv.26018
- WHO. COVID-19 Weekly Epidemiological Update 48 [Internet]. World Health Organization. 2021. p. 1–16. Disponível em: https://www.who.int/publications/m/item/weekly-epidemiological-update-on-covid-19—13-july-2021
- Candido DS, Claro IM, de Jesus JG, Souza WM, Moreira FRR, Dellicour S, et al. Evolution and epidemic spread of SARS-CoV-2 in Brazil. Science (80- ) [Internet]. 2020 Sep 4;369(6508):1255–60. Disponível em: https://www.sciencemag.org/lookup/doi/10.1126/science.abd2161
- Supasa P, Zhou D, Dejnirattisai W, Liu C, Mentzer AJ, Ginn HM, et al. Reduced neutralization of SARS-CoV-2 B.1.1.7 variant by convalescent and vaccine sera. Cell. 2021;184(8):2201-2211.e7. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7891044/
- Cerutti G, Guo Y, Zhou T, Gorman J, Lee M, Rapp M, et al. Potent SARS-CoV-2 neutralizing antibodies directed against spike N-terminal domain target a single supersite. Cell Host Microbe [Internet]. 2021 Mar; Disponível em: https://linkinghub.elsevier.com/retrieve/pii/S1931312821001335
- Bal A, Destras G, Gaymard A, Stefic K, Marlet J, Eymieux S, et al. Two-step strategy for the identification of SARS-CoV-2 variant of concern 202012/01 and other variants with spike deletion H69-V70, France, August to December 2020. Euro Surveill [Internet]. 2021;26(3). Disponível em: http://www.ncbi.nlm.nih.gov/pubmed/33478625
- Davies NG, Abbott S, Barnard RC, Jarvis CI, Kucharski AJ, Munday JD, et al. Estimated transmissibility and impact of SARS-CoV-2 lineage B.1.1.7 in England. Science (80- ) [Internet]. 2021 Apr 9;372(6538):eabg3055. Disponível em: https://www.sciencemag.org/lookup/doi/10.1126/science.abg3055
- Challen R, Brooks-Pollock E, Read JM, Dyson L, Tsaneva-Atanasova K, Danon L. Risk of mortality in patients infected with SARS-CoV-2 variant of concern 202012/1: matched cohort study. BMJ [Internet]. 2021;372:n579. Disponível em: http://www.ncbi.nlm.nih.gov/pubmed/33687922
- Davies NG, Jarvis CI, Edmunds WJ, Jewell NP, Diaz-Ordaz K, Keogh RH. Increased mortality in community-tested cases of SARS-CoV-2 lineage B.1.1.7. Nature [Internet]. 2021 Mar 15; Disponível em: http://www.nature.com/articles/s41586-021-03426-1
- Brookman S, Cook J, Zucherman M, Broughton S, Harman K, Gupta A. Effect of the new SARS-CoV-2 variant B.1.1.7 on children and young people. Lancet Child Adolesc Heal [Internet]. 2021;5(4):e9–10. Disponível em: http://dx.doi.org/10.1016/S2352-4642(21)00030-4
- Funk T, Pharris A, Spiteri G, Bundle N, Melidou A, Carr M, et al. Characteristics of SARS-CoV-2 variants of concern B.1.1.7, B.1.351 or P.1: data from seven EU/EEA countries, weeks 38/2020 to 10/2021. Euro Surveill [Internet]. 2021;26(16):1–10. Disponível em: http://www.ncbi.nlm.nih.gov/pubmed/33890566
- Gómez CE, Perdiguero B, Esteban M. Emerging sars-cov-2 variants and impact in global vaccination programs against sars-cov-2/covid-19. Vaccines. 2021;9(3):1–13.
- Slavov SN, Patané JSL, Bezerra R dos S, Giovanetti M, Fonseca V, Martins AJ, et al. Genomic monitoring unveil the early detection of the SARS-CoV-2 B.1.351 lineage (20H/501Y.V2) in Brazil. medRxiv [Internet]. 2021;1–17. Disponível em: https://www.medrxiv.org/content/10.1101/2021.03.30.21254591v1
- Nelson G, Buzko O, Spilman P, Niazi K, Rabizadeh S, Soon-Shiong P. Molecular dynamic simulation reveals E484K mutation enhances spike RBD-ACE2 affinity and the 2 combination of E484K, K417N and N501Y mutations (501Y.V2 variant) induces conformational change greater than N501Y mutant alone, potentially resulting in an escape mutant. bioRxiv [Internet]. 2021; Disponível em: https://www.biorxiv.org/content/10.1101/2021.01.13.426558v1.full.pdf
- Wang P, Casner RG, Nair MS, Wang M, Yu J, Liu L, et al. Increased Resistance of SARS-CoV-2 Variant P . 1 to Antibody Neutralization. Cell Host Microbe [Internet]. 2021 May 12;29(5):747-751.e4.; Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8053237/pdf/main.pdf
- Cele S, Gazy I, Jackson L, Hwa SH, Tegally H, Lustig G, et al. Escape of SARS-CoV-2 501Y.V2 from neutralization by convalescent plasma. Nature. 2021 Mar 593(7857) 142-146; Disponível em: https://doi.org/10.1038/s41586-021-03471-w
- Zhou D, Dejnirattisai W, Supasa P, Liu C, Mentzer AJ, Ginn HM, et al. Evidence of escape of SARS-CoV-2 variant B.1.351 from natural and vaccine-induced sera. Cell [Internet]. 2021 Apr;184(9):2348-2361.e6. Disponível em: https://linkinghub.elsevier.com/retrieve/pii/S0092867421002269
- Science Brief: Emerging SARS-CoV-2 Variants [Internet]. Centers for Disease Control and Prevention – CDC; 2021. Disponível em: https://www.cdc.gov/coronavirus/2019-ncov/science/science-briefs/scientific-brief-emerging-variants.html
- Faria N, Claro IM, Candido D, Franco LAM, Andrade PS, Coletti TM, et al. Genomic characterisation of an emergent SARS-CoV-2 lineage in Manaus: preliminary findings. Virological Prepr [Internet] 2021; Disponível em: https://virological.org
- Wang Z, Schmidt F, Weisblum Y, Muecksch F, Barnes CO, Finkin S, et al. mRNA vaccine-elicited antibodies to SARS-CoV-2 and circulating variants. Nature [Internet]. 2021;592(April). Disponível em: http://dx.doi.org/10.1038/s41586-021-03324-6
- Freitas ARR, Beckedorff OA, Cavalcanti LP, Siqueira AM, Castro DB de, Costa CF da. The emergence of novel SARS-CoV-2 variant P.1 in Amazonas (Brazil) was temporally associated with a change in the age and gender profile of COVID-19 mortality. Scielo Prepr [Internet]. 2021; Disponível em: https://preprints.scielo.org/index.php/scielo/preprint/view/2030
- Voloch CM, da Silva Francisco R, de Almeida LGP, Cardoso CC, Brustolini OJ, Gerber AL, et al. Genomic characterization of a novel SARS-CoV-2 lineage from Rio de Janeiro, Brazil. J Virol [Internet]. 2021 Mar 1; Disponível em: http://www.ncbi.nlm.nih.gov/pubmed/33649194
- Baum A, Fulton BO, Wloga E, Copin R, Pascal KE, Russo V, et al. Antibody cocktail to SARS-CoV-2 spike protein prevents rapid mutational escape seen with individual antibodies. Science. 2020 Jun; 369(6506):1014-8. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7299283
- Greaney AJ, Loes AN, Crawford KHD, Starr TN, Malone KD, Chu HY, et al. Comprehensive mapping of mutations in the SARS-CoV-2 receptor-binding domain that affect recognition by polyclonal human plasma antibodies. Cell Host Microbe [Internet]. 2021;29(3):463-476.e6. Disponível em: https://doi.org/10.1016/j.chom.2021.02.003
- Liu P, Cai J, Jia R, Xia S, Wang X, Cao L, et al. Dynamic surveillance of SARS-CoV-2 shedding and neutralizing antibody in children with COVID-19. Emerg Microbes Infect [Internet]. 2020 Jan 1;9(1):1254–8. Disponível em: https://www.tandfonline.com/doi/full/10.1080/22221751.2020.1772677
- Nonaka CK V., Franco MM, Gräf T, de Lorenzo Barcia CA, de Ávila Mendonça RN, de Sousa KAF, et al. Genomic Evidence of SARS-CoV-2 Reinfection Involving E484K Spike Mutation, Brazil. Emerg Infect Dis [Internet]. 2021 May;27(5):1522-4. Disponível em: https://wwwnc.cdc.gov/eid/article/27/5/21-0191_article.htm
- Fintelman-Rodrigues N, Silva AP da, Santos MC dos, Saraiva FB, Ferreira MA, Gesto J, et al. Genetic Evidence and Host Immune Response in Persons Reinfected with. Emerg Infect Dis [Internet]. 2021;27(5):1-12. Disponível em: https://wwwnc.cdc.gov/eid/article/27/5/20-4912_article
- Cherian S, Potdar V, Jadhav S, Yadav P, Gupta N, Das M, et al. Convergent evolution of SARS-CoV-2 spike mutations, L452R, E484Q and P681R, in the second wave of COVID-19 in Maharashtra, India. bioRxiv [Internet]. 2021;2021.04.22.440932. Disponível em: https://doi.org/10.1101/2021.04.22.440932
- Menezes M. Portal Fiocruz [Internet]. 2021. Disponível em: https://portal.fiocruz.br/noticia/pesquisa-sugere-maior-risco-de-reinfeccao-pela-variante-delta
- Edara V-V, Lai L, Sahoo MK, Floyd K, Sibai M, et al. Infection and vaccine-induced neutralizing antibody responses to the SARS-CoV-2 B.1.617.1 variant. N Engl J Med [Internet] 2021 Aug; 385:664-666. Disponível em: https://www.nejm.org/doi/full/10.1056/NEJMc2107799
- Liu C, Ginn HM, Dejnirattisai W, Ren J, Stuart DI, Screaton GR. Article Reduced neutralization of SARS-CoV-2 B . 1 . 617 by vaccine and convalescent serum Reduced neutralization of SARS-CoV-2 B. 1 . 617 by vaccine and convalescent serum. Cell. 2021;184:1–17.
- Romero EP, Dávila-Barclay A, Salvatierra G, Gonzáles L, Cuicapuza D, et al. The Emergence of SARS-CoV-2 Variant Lambda (C.37) in South America. medRxiv [Internet]. 2021; Disponível em: https://www.medrxiv.org/content/10.1101/2021.06.26.21259487v1
- Wink LP, Volpato FCZ, Monteiro FL, Willig JB, Zavascki AP, et al. First identification of SARS-CoV-2 Lambda (C.37) variant in Southern Brazil. medRxiv [Internet]. 2021; Disponível em: https://www.medrxiv.org/content/10.1101/2021.06.21.21259241v1
- Acevedo ML, Palomares LA-, Bustamante A, Gaggero A, Paredes F, et al. Infectivity and immune escape of the new SARS-CoV-2 variant of interest Lambda. medRxiv [Internet]. 2021; Disponível em: https://www.medrxiv.org/content/10.1101/2021.06.28.21259673v1
- Resende PC, Gräf T, Paixão ACD, Appolinario L, Lopes RS, Mendonça AC da F, et al. A Potential SARS-CoV-2 Variant of Interest (VOI) Harboring Mutation E484K in the Spike Protein Was Identified within Lineage B.1.1.33 Circulating in Brazil. Viruses [Internet]. 2021 Apr 21;13(5):724. Disponível em: https://www.mdpi.com/1999-4915/13/5/724
- Lamarca AP, Almeida LGP, Francisco Jr R da S, Lima LFA, Scortecci KC, Perez VP, et al. Genomic surveillance of SARS-CoV-2 tracks early interstate transmission of P.1 lineage and diversification within P.2 clade in Brazil. medRxiv [Internet]. 2021; Disponível em: https://www.medrxiv.org/content/10.1101/2021.03.21.21253418v2.full
- Resende PC, Gräf T, Neto LGL, Silva FV da, Paixão ACD, Appolinario L, et al. Identification of a new B.1.1.33 SARS-CoV-2 Variant of Interest (VOI) circulating in Brazil with mutation E484K and multiple deletions in the amino (N)-terminal domain of the Spike protein. Virological [Internet]. 2021; Disponível em: https://virological.org/t/identification-of-a-new-b-1-1-33-sars-cov-2-variant-of-interest-voi-circulating-in-brazil-with-mutation-e484k-and-multiple-deletions-in-the-amino-n-terminal-domain-of-the-spike-protein/675
- Francisco Jr R da S, Benites LF, Lamarca AP, de Almeida LGP, Hansen AW, Gularte JS, et al. Pervasive transmission of E484K and emergence of VUI-NP13L with evidence of SARS-CoV-2 co-infection events by two different lineages in Rio Grande do Sul, Brazil. Virus Res [Internet]. 2021 Apr;296:198345. Disponível em: https://linkinghub.elsevier.com/retrieve/pii/S0168170221000526
- P4 a mais nova variante identificada no Brasil [Internet]. Sociedade Brasileira de Virologia. 2021. Disponível em: https://sbv.org.br/sbv/p-4-a-mais-nova-variante-identificada-no-brasil/
Correspondência
Cleonice Maria Michelon
Departamento de Análises Clínicas – ACL,
Centro de Ciências da Saúde – CCS,
Universidade Federal de Santa Catarina – UFSC,
Campus Universitário Reitor João David Ferreira Lima,
Trindade – Florianópolis – CEP 88 040-900
Tel: (48) 3721-4159 – E-mail: [email protected]
