Avaliação multicêntrica de um método livre de purificação para detecção do vírus SARS-CoV-2
Multicentric evaluation of a free purification method for detecting SARS-CoV-2 virus
Rafael Oliveira Reis1, Leandro Bolzan2, Cintia Lima Ambrozio1, Guilherme Cassao Bragança3, Gustavo Dupont Garrastazu3
1 Centro Universitário URCAMP, Laboratório de Biologia Molecular. Bagé, RS. Brasil.
2 UNIFRA – Centro Universitário Franciscano. Santa Maria, RS, Brasil.
3 Centro Universitário URCAMP, Curso de Farmácia. Bagé, RS, Brasil.
Recebido em 10/03/2022
Aprovado em 24/10/2022
DOI: 10.21877/2448-3877.202200030
INTRODUÇÃO
No final de 2019, um surto de pneumonia de etiologia desconhecida ocorrido na cidade de Wuhan, província de Hubei na China, foi relatado pela Organização Mundial de Saúde (OMS). Um novo tipo de Coronavírus, denominado como SARS-CoV-2, levou à morte de milhares de indivíduos no mundo todo.(1,2) A volta à rotina com segurança tem sido estudada e vem avançando no mundo todo, entretanto novas variantes preocupantes do vírus SARS-CoV-2 têm comumente emergido.(3,4) A massiva testagem mundial para identificar indivíduos infectados pelo SARS-CoV-2 produziu grande impacto nos laboratórios de diagnóstico, levando a momentos críticos diante da grande escassez de insumos plásticos e de reagentes.(5)
No atual momento, com a retomada das atividades em vários países, abertura de fronteiras, grandes eventos presenciais, a testagem massiva com testes de alta sensibilidade se mostrou ainda mais fundamental. Para isso são necessários testes com tempos de resposta rápida com o intuito de diminuir ou evitar novas ondas e surtos de Covid-19, e assim bloquear o avanço de novas variantes preocupantes.(4,6,7)
Novas formas diagnósticas para identificação do vírus são sugeridas na literatura, com inovações que visam aumentar a escala de testagem e no menor tempo possível.(8) Entretanto, a precisão dos métodos moleculares é fundamental para a eficiência na detecção do vírus SARS-CoV-2. A técnica de RT-PCR livre de purificação ou direta tem sido descrita e testada como alternativa rápida através da técnica RT-qPCR. O método QiaPrep&Amp Viral Um Kit® (QP&A – Hilden, Alemanha), tem por objetivo disponibilizar resultados rápidos utilizando protocolo de reação direta sem a necessidade de purificação de ácidos nucleicos, através de somente uma etapa de lise de amostra. Sendo assim, o trabalho focou em analisar o desempenho do método QP&A em dois laboratórios que realizam a testagem para SARS-COV-2 frente ao método XPert XPress SARS-CoV-2® (XP SARS-CoV-2, CEPHEID – Califórnia, EUA).
METODOLOGIA
Obtenção das Amostras
Uma análise multicêntrica foi realizada em dois laboratórios da Região Sul do Brasil. Foram selecionadas aleatoriamente 85 amostras com resultados detectáveis e não detectáveis para SARS-CoV-2 previamente processadas no equipamento Gene Xpert (Califórnia – EUA) utilizando o teste IVD (diagnostic in vitro) XP SARS-CoV-2. No LAB “A” foram avaliadas 45 amostras, sendo 35 detectáveis para SARS-CoV-2 e 10 não detectáveis; no LAB “B” foram realizadas as análises de 40 amostras (25 detectáveis e 15 não detectáveis).
Análises das Amostras QP&A
As amostras com resultados detectáveis foram armazenadas a -30°C em meio de transporte viral (VTM) (CEPHEID -EUA), para o estudo. As amostras foram deixadas em temperatura ambiente em cabine de segurança biológica até completo descongelamento. Em seguida, foram submetidas ao método QP&A. Para a reação foram utilizados 8µL de amostra em VTM e adicionados 2µL do tampão de lise do kit, e incubando em TA (temperatura ambiente) por 2 minutos. Após, foram adicionados o MIX previamente preparada com 5µL de Mastermix, 2µL de primers e sondas do controle interno de amplificação, 1,0ul do de pirmers e sondas do controle interno de amostras, 1,0 de primers e sondas para o gene N1/N2 e 1,0µL de água livre de DNAse e RNAse por reação, somando total de 10µL de PCR por reação, que posteriormente foi acrescentado à amostra com tampão de lise com volume total de reação de 10µL.
Todas as amostras selecionadas foram testadas em equipamento QiaQuant 96/5 plex, utilizando o kit comercial QP&A e sondas e primers (QIAGEN N1+N2 Assay, Hilden – ALE) descritas no protocolo CDC. As condições RT-PCR foram de 10 minutos a 50°C para formação de DNA, seguidos por extração inicial de 2 minutos para DNA genômico. A formação de amplicons foi a realizada através de 40 ciclos de extração a 95°C por 5 segundos, seguidos por anelamento, com extensão e leitura a 58°C por 30 segundos.
A relação entre os valores de cycle threshold (Ct) obtidos das amostras detectáveis para o gene N1/N2 do kit QP&A foi comparada à dos valores do gene N2 do kit XP SARS-CoV-2. As condições de amplificação e protocolos seguiram as regras do fabricante.
Avaliação de Desempenho
Para a avaliação da metodologia em diferentes cenários epidemiológico, o valor preditivo positivo (VPP) e o valor preditivo negativo (VPN), assim como a Taxa de probabilidade (Likelihood ratio – LR) foram avaliados frente a três cenários hipotéticos de prevalência de 1%, 10% e 25%.
RESULTADOS
Do total de 85 amostras analisadas (60 detectáveis e 25 não detectáveis), 59 foram detectáveis para QP&A e 24 não detectáveis, sendo que as duas amostras restantes apresentaram resultados divergentes entre os dois métodos testados. Do total de amostras obtivemos um resultado falso positivo e um resultado falso negativo; em comparação com o método utilizado como referência XP, ambas as discrepâncias ocorreram no LAB “B”. Os Cts das amostras positivas para o gene viral variaram de 12,8 ciclos a 36,60 ciclos para o XP SARS-CoV-2 e 14,60 ciclos a 38,60 ciclos para o QP&A (Figura 1). Os dados foram submetidos ao teste de normalidade de quando analisamos a diferença entre as médias de Ct entre os dois grupos, no qual não houve diferença significativa entre os dois testes, p>0,005.
Não houve diferença significativa entre os Cts encontrados pelo método XP SARS-CoV-2 e o método QP&A. A Figura 2 demonstra as amostras que apresentaram maior diferença entre os Cts (15, 30, 34 e 35). Através da análise de correlação bivariada foi possível verificar a boa correlação entre os dois métodos R=0.932, (IC-95%) (Figura 3).
Ficou evidente que o método apresenta melhor sensibilidade quando em amostras com cargas virais mais elevadas Ct <30 ciclos, entretanto apenas uma amostra com Ct acima de 30 apresentou resultado falso negativo (amostra com Ct de 36,9 no XP SARS-CoV-2), conforme Tabela 1.
Encontramos uma sensibilidade total de 98,3% (IC 95% 91,06 – 99,96%) e uma especificidade de 96% (IC 79,65% – 99,9 %), com acurácia total de 97,5%. Porém quando avaliamos amostras com Ct até 35 ciclos obtidos pelo método referência, a concordância entre as amostras positivas foi de 100%.
As duas amostras com resultados discordantes foram repetidas em RT-qPCR (dados não apresentados) através do kit (Xgen Master Covid-19 – Mobius Life Science), onde foi demonstrado que o resultado falso positivo observado se tratava de um resultado verdadeiramente positivo, e com isso pudemos observar uma especificidade de 100%.
Para avaliar o impacto da nova metodologia frente aos cenários pandêmicos, inferimos a análise em três tipos de prevalência, sendo a primeira 1% de positividade, seguida por 10% e 25% de positividade.
Inicialmente a uma baixa taxa de infecção na população 1%, a taxa de probabilidade (Likelihood ratio- LR) que indica quantas vezes um resultado diagnóstico é capaz de mudar a probabilidade de ser uma doença, sendo calculado para resultados positivos e negativos, LR + foi de 24,58 (IC 3,60 – 167,80) enquanto a razão de probabilidade negativa LR- foi de 0,02 IC (0- 0,02). Já o valor preditivo positivo (VPP) foi 19,89% (IC 3,51% -62,89%), enquanto o valor preditivo negativo (VPN) do teste neste cenário foi de 99,98% (99,88% – 100%).
Quando modificamos a prevalência da doença para 10%, LR+ é de 24,58, (3,60 – 167,80), ao mesmo tempo em que o LR é de 0,02 (IC 0 – 0,02). O VPP foi de 73,2% (IC 28,58% – 94,1%), de forma que o VPN encontrado foi 99,81% (IC 98,67% – 99,97%).
Em um cenário de forte prevalência da doença supondo uma taxa de prevalência de 25%, observamos um LR+ de 24,58 (3,60 – 167,80), enquanto a taxa de probabilidade negativa foi igual à dos outros dois cenários LR- 0,02 IC (0 – 0,02). Já o valor preditivo positivo do teste em um cenário de alta prevalência foi de 89,12% (IC 54,56% – 98,24%), enquanto o valor preditivo negativo se manteve por volta do 99% (IC 96,1% – 99,92).
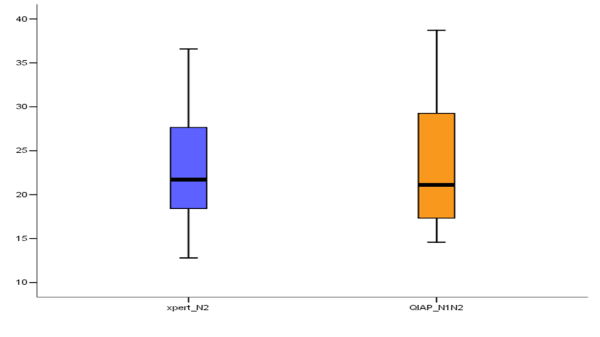
Figura 1
Média dos Cts observados entre os dois métodos
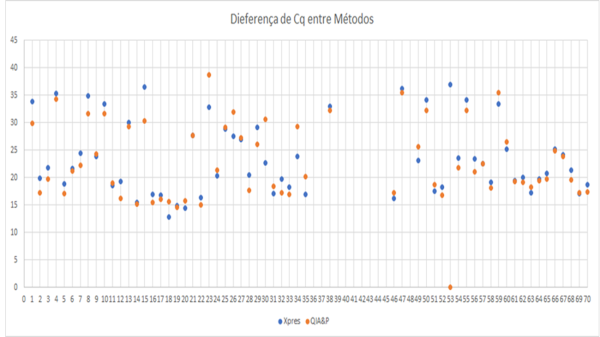
Figura 2
Diferença de Ct entre as amostras testadas no XP SARS-CoV-2 (azul) e no QP&A (laranja).
Legenda: Cq – ciclo de qualificação (corresponde a Ct)
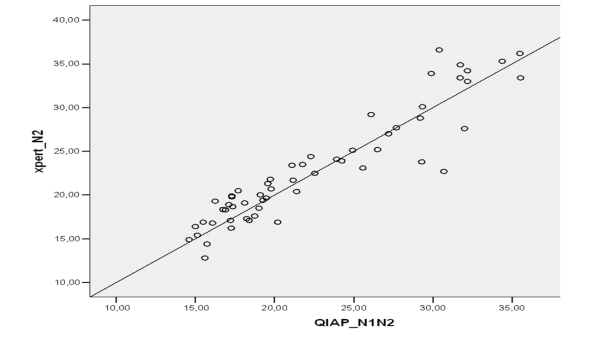
Figura 3
Análise de regressão linear bivariada para avaliar relação entre os dois métodos.
Tabela 1
Avaliação da concordância em diferentes grupos de Cts.
| Xpert Xpress SARS-CoV-2 | QIA&P | Concordância | Erros | |
| N Pos | N | % | Falso Neg Ct | |
| Pos <30 Ct | 47 | 47 | 100 | * |
| Pos >30 Ct | 13 | 12 | 92,30 | 36,9 |
| Falso Pos Ct | ||||
| Negativo | 25 | 24 | 96 | 38,3 |
DISCUSSÃO
O teste RT-PCR tem sido amplamente empregado no diagnóstico da Covid-19, devido ao elevado gasto de insumos plásticos aliado ao elevado tempo de execução do teste RT-qPCR convencional. Com isso, a inovação para melhorar estes aspectos tem sido fomentada.(5)
A técnica de PCR quantitativa (qPCR) para a detecção de um RNA de interesse é precedida do isolamento e purificação do RNA total da amostra, da eluição e possível concentração do material.(8) Só então é utilizado o RNA purificado para reação de RT-qPCR, e sendo assim a obtenção do ácido nucleico é trabalhosa e demorada.(9) Vale considerar que etapas adicionais que requerem manipulação manual podem resultar em erros experimentais.(10)
Além disso, o uso de materiais plásticos em grandes quantidades para extração de ácidos nucleicos pode resultar em falta nos estoques, podendo levar à interrupção do serviço devido à falta de insumos. Assim, os métodos de extração direta e livre de purificação tornaram-se uma alternativa, pois envolvem menos etapas no processo e possuem baixo consumo de insumos plásticos (escassos em momentos pandêmicos).(11-14)
Vários autores têm explorado esta necessidade do sistema diagnóstico,(15,16) no entanto nem todas as metodologias apresentam ótima sensibilidade.
Pessoa et al. (2021) obtiveram uma concordância de 78% entre amostras positivas com uma diferença significativa no valor de Ct quando comparadas ao método padrão no total de amostras do estudo. O método QP&A apresentou melhor acurácia, quando comparado ao método realizado por Pessoa et al. (2021).
Claas et al. (2021), utilizando o mesmo método e protocolo, obtiveram alta concordância (100%) entre seus resultados quando avaliados em amostras com detecção para o gene N1/N2 demonstrando a reprodutibilidade do método. Quando o método livre de purificação foi comparado a quatro plataformas diferentes de RT-PCR, avaliando resultados com todos os Cts, a concordância do autor foi de 86,3%. Q&AP demonstrou boa sensibilidade clínica em nosso estudo, quando comparado ao kit já validado Xpress-SARS-CoV-2, avaliando amostras detectáveis com Ct até 40 ciclos, realizados no equipamento Gene Xpert. O método Q&AP também apresentou desempenho superior ao do encontrado por Pessoa et al. (2021), com a utilização do método in house rápido de obtenção de ácidos nucleicos.
O método Q&AP apresentou seu melhor desempenho em amostras detectáveis com Ct até 35 ciclos. Outras alternativas, conforme Smyrlaki et al. (2020), através de detecção e aquecimento para otimizar a detecção molecular da Covid-19, obtiveram sensibilidade de 96% e 91% em dois diferentes protocolos utilizando amostras para o gene ORF1ab até Ct de 40 ciclos, avaliando 597 amostras. Visseaux et al. (2021), avaliando métodos rápidos e diretos, obtiveram sensibilidade de 91,9%, 89,2% e 94,6% em amostras com Ct até 30 ciclos, utilizando kits com alvos para o gene E dos SARS-CoV-2.(17,18)
QP&A apresentou ótimo desempenho quando foram avaliadas amostras com Ct até 35 ciclos, sendo superior a outros estudos.(11,16) O método QP&A apresentou sensibilidade de 100% com a avaliação até 30 Cts, demonstrando desempenho semelhante ao dos métodos disponíveis no mercado laboratorial.(11,13,14)
Dois resultados discrepantes foram encontrados no QP&A em nosso estudo, também observados em outros estudos utilizando o mesmo equipamento como referência.(5) Porém quando avaliamos a sensibilidade e especificidade associadas a valores preditivos, verificamos um excelente valor preditivo negativo nos diversificados cenários de prevalência pela infecção da Covid-19. Ondas de disseminação do vírus intercaladas por períodos de baixa disseminação têm sido constantes no mundo todo.(19) A simulação dos variados cenários contribui para o entendimento do real desempenho clínico do método em períodos de alta disseminação e períodos de baixa transmissão do vírus. Como esperado, o valor preditivo do teste foi melhor quando em elevadas taxas de prevalência da doença, demonstrando tanto em cenários de baixa prevalência que o teste ainda apresenta desempenho aceitável. Além disso, o aumento da capacidade de testagem nos diferentes tipos de laboratórios pode contribuir para um diagnóstico rápido e mais preciso do que testes que utilizam de fluxo lateral, principalmente em contatos e indivíduos assintomáticos.(20,21)
CONCLUSÃO
O método RUO QP&A apresentou boa acurácia quando comparado ao método utilizado como referência, a elevada especificidade associada a uma boa sensibilidade. A implementação deste teste pode contribuir fortemente para auxiliar no combate à pandemia da Covid-19, principalmente no momento de retomada das atividades. O menor tempo para realização do teste associado à menor utilização de insumos plásticos pode auxiliar na velocidade de isolamento de pacientes positivos, contribuindo para a diminuição de variantes de preocupação, e é importante ferramenta no controle e disseminação do vírus SARS-CoV-2, principalmente no momento de reabertura das fronteiras no mundo todo. Avaliações com maior número amostral pode contribuir para determinar ainda melhor este desempenho e o impacto no controle pandêmico.
REFERÊNCIAS
- Matta S, Chopra KK, Arora VK. Morbidity and mortality trends of Covid 19 in top 10 countries. Indian J Tuberc. 2020;67(4):S167. doi:10.1016/J.IJTB.2020.09.031
- Guan W, Ni Z, Hu Y, et al. Clinical Characteristics of Coronavirus Disease 2019 in China. N Engl J Med. 2020;382(18):1708–1720. doi:10.1056/nejmoa2002032
- Hierro LA, Cantarero D, Patiño D, de Arenaza DRP. Who can go back to work when the COVID-19 pandemic remits? PLoS One. 2020;15(8). doi:10.1371/JOURNAL.PONE.0238299
- Gao S, Guo H, Luo G. Omicron variant (B.1.1.529) of SARS-CoV-2, a global urgent public health alert! J Med Virol. novembro 2021. doi:10.1002/JMV.27491
- Becker M, Taylor T, Kiazyk S, Cabiles D, Meyers A, Sandstrom P. Recommendations for sample pooling on the Cepheid GeneXpert ® system using the Cepheid Xpert ® Xpress SARS-CoV-2 assay. 2020:1-13. doi:10.1101/2020.05.14.097287
- Das S, Shibib DR, Vernon MO. Since January 2020 Elsevier has created a COVID-19 resource centre with free information in English and Mandarin on the novel coronavirus COVID- 19 . The COVID-19 resource centre is hosted on Elsevier Connect , the company ’ s public news and information. 2020;(January).
- Tegally H, Wilkinson E, Lessells RJ, et al. Sixteen novel lineages of SARS-CoV-2 in South Africa. Nat Med. fevereiro 2021. doi:10.1038/s41591-021-01255-3
- Alcoba-Florez J, González-Montelongo R, Íñigo-Campos A, et al. Fast SARS-CoV-2 detection by RT-qPCR in preheated nasopharyngeal swab samples. Int J Infect Dis. 2020;97:66–68. doi:10.1016/J.IJID.2020.05.099
- Menezes ME, Lima LM, Martinello F. Diagnóstico laboratorial do SARS-CoV-2 por transcrição reversa seguida de reação em cadeia da polimerase em tempo real (RT-PCR). Rev Bras Análises Clínicas. 2020;52(2). doi:10.21877/2448-3877.20200006
- Arevalo-Rodriguez I, Buitrago-Garcia D, Simancas-Racines D, et al. False-negative results of initial RT-PCR assays for COVID-19: A systematic review. PLoS One. 2020;15(12). doi:10.1371/JOURNAL.PONE.0242958
- Bruce EA, Huang ML, Perchetti GA, et al. Direct RT-qPCR detection of SARS-CoV-2 RNA from patient nasopharyngeal swabs without an RNA extraction step. PLoS Biol. 2020;18(10):e3000896. Published 2020 Oct 2. doi:10.1371/journal.pbio.3000896
- Hasan MR, Mirza F, Al-Hail H, et al. Detection of SARS-CoV-2 RNA by direct RT-qPCR on nasopharyngeal specimens without extraction of viral RNA. PLoS One. 2020;15(7 July). doi:10.1371/JOURNAL.PONE.0236564
- Merindol N, Pépin G, Marchand C, et al. SARS-CoV-2 detection by direct rRT-PCR without RNA extraction. J Clin Virol. 2020;128. doi:10.1016/J.JCV.2020.104423
- Barza R, Patel P, Sabatini L, Singh K. Use of a simplified sample processing step without RNA extraction for direct SARS-CoV-2 RT-PCR detection. J Clin Virol. 2020;132. doi:10.1016/J.JCV.2020.104587
- Claas ECJ, Smit PW, van Bussel MJAWM, et al. A two minute liquid based sample preparation for rapid SARS-CoV2 real-time PCR screening: A multicentre evaluation. J Clin Virol. 2021;135:104720. doi:10.1016/J.JCV.2020.104720
- Pessoa WFB, Galvão BHA, Barroso H, et al. Alternative workflow for COVID-19 diagnosis using direct RT-PCR screening. Rev Bras Análises Clínicas. 2021;53(2):175–179. doi:10.21877/2448-3877.202102128
- Smyrlaki I, Ekman M, Lentini A, et al. Massive and rapid COVID-19 testing is feasible by extraction-free SARS-CoV-2 RT-PCR. Nat Commun. 2020;11(1):1–12. doi:10.1038/s41467-020-18611-5
- Visseaux B, Collin G, Houhou-Fidouh N, et al. Evaluation of three extraction-free SARS-CoV-2 RT-PCR assays: A feasible alternative approach with low technical requirements. J Virol Methods. 2021;291:114086. doi:10.1016/j.jviromet.2021.114086
- Seong H, Hyun HJ, Yun JG, et al. Comparison of the second and third waves of the COVID-19 pandemic in South Korea: Importance of early public health intervention. Int J Infect Dis. 2021;104:742. doi:10.1016/J.IJID.2021.02.004
- Rodrigues Teixeira Silva Daameche H, Zampieri Ipolito M, Chieregato Matheus JP. Indicadores de Lesão e Inflamação Muscular em Atletas – Revisão Integrativa da Literatura. Rev Educ em Saúde. 2018;6(1):90-98. doi:10.29237/2358-9868.2018v6i1.p90-98
- Torres I, Poujois S, Albert E, Colomina J, Navarro D. Evaluation of a rapid antigen test (PanbioTM COVID-19 Ag rapid test device) for SARS-CoV-2 detection in asymptomatic close contacts of COVID-19 patients. Clin Microbiol Infect. 2021;27(4):636.e1-636.e4. doi:10.1016/J.CMI.2020.12.022
Correspondência
Rafael Oliveira Reis
E-mail: [email protected]
