Staphylococcus lugdunensis em hemoculturas: perfil de susceptibilidade aos antimicrobianos
Staphylococcus lugdunensis in blood cultures: susceptibility profile of antimicrobials
Danielle Guimarães Araújo1
Marcelo Eduardo Ferreira Oliveira2
Sibele Ribeiro de Oliveira3
1 Biomédica. Graduada pelo Centro Universitário Tabosa de Almeida Asces-Unita – Caruaru-PE, Brasil.
2 Graduado. Centro Universitário Tabosa de Almeida Asces-Unita – Caruaru-PE, Brasil.
3 Doutora em Ciências Biológicas – Universidade Federal de Pernambuco – UFPE – Recife-PE, Brasil.
Instituição: Centro Universitário Tabosa de Almeida Asces-Unita – Caruaru-PE, Brasil.
Conflito de interesses: não há conflito de interesses
Artigo recebido em 03/07/2017
Artigo aprovado em 18/10/2017
DOI: 10.21877/2448-3877.201700592
Resumo
Objetivo: Determinar o perfil de sensibilidade e resistência de antimicrobianos frente a Staphylococcus lugdunensis isolados de hemoculturas de um hospital de emergência. Métodos: Amostras de hemocultura foram aspiradas e semeadas no meio de cultura ágar Sangue de Carneiro. Foi realizado o teste de catalase para identificação do gênero Staphylococcus sp. e, para identificação fenotípica de espécies de interesse clínico, as amostras foram submetidas aos testes de DNase, Novobiocina e PYR. Para análise do perfil de sensibilidade e resistência, o antibiograma foi utilizado. Resultados: Das 95 hemoculturas analisadas, 92% mostraram crescimento bacteriano. Destas, 6% foram identificadas como Staphylococcus lugdunensis, mostrando-se resistentes a cefoxitina, eritromicina, clindamicina e tetraciclina. A sensibilidade ocorreu frente à nitrofurantoína e linezolida. Conclusão: O achado de Staphylococcus lugdunensis foi um dado relevante, uma vez que trabalhos envolvendo esta espécie são escassos. O S. lugdunensis é um patógeno emergente, com elevado potencial agressor. Dessa forma, sua correta identificação laboratorial, bem como o monitoramento do perfil de susceptibilidade e resistência, podem contribuir com um melhor direcionamento de medidas de controle da infecção, principalmente em hospitais.
Palavras-chave
Staphylococcus lugdunensis; Infecção hospitalar; Antibacterianos
INTRODUÇÃO
Os Staphylococcus coagulase negativa (SCN) estão entre os microrganismos Gram positivos mais frequentemente isolados no laboratório de microbiologia, fazendo parte da microbiota normal da pele e sendo muitas vezes considerados como colonizadores quando isolados a partir de amostras clínicas.(1,2) Seu significado clínico é difícil de se estabelecer, uma vez que são naturais da pele e membranas mucosas e têm sido considerados patógenos inócuos ou oportunistas de virulência variada. As infecções causadas por SCN, ao contrário daquelas produzidas por Staphylococcus aureus, manifestam-se como doenças menos graves ou subagudas, mais raramente associadas à mortalidade.(2)
O Staphylococcus lugdunensis, mesmo fazendo parte do grupo dos SCN, tem significativo potencial de virulência.(1,3) Desde que foi primeiro descrito em 1988, o S. lugdunensis vem sendo associado a casos de endocardite, infecção da pele, peritonite, abcesso cerebral, osteomielite, infecção vascular protética e septicemia, sendo as manifestações de pele e tecidos moles as mais comuns.(4-8) Sua patogenia é similar à do S. aureus pela presença de fatores de virulência em comum. Quanto aos baixos percentuais de isolamento desta bactéria, estes podem estar ligados ao fato de compartilharem semelhanças fenotípicas com S. aureus, o que pode dificultar a sua identificação.(9-11)
Esta limitação se deve ao fato de um percentual variável de isolados de S. lugdunensis reagir a testes rápidos de identificação, como a pesquisa do clumping factor, sendo positivos quando realizados os testes de coagulase em lâmina ou de aglutinação em látex. Por essa razão, outros testes laboratoriais fenotípicos são necessários para confirmar sua presença, como o teste de Pyrrolidonil arilamidase (PYR).(8,10-12)
O S. lugdunensis é ainda descrito como produtor de biofilme, uma matriz extracelular polimérica que facilita a adesão em superfícies, sendo que tal habilidade promove proteção contra a resposta imune do hospedeiro, bem como pode proporcionar maior resistência às doses de antimicrobianos normalmente utilizados.(9,10) Alguns relatos afirmam a produção de beta-lactamases por este patógeno, o que lhe confere resistência bacteriana significativa e maior importância clínica.(4,9-11)
Vale ressaltar que, em alguns casos, o S. lugdunensis pode apenas representar a contaminação da hemocultura; entretanto, a correta distinção entre os S. lugdunensis e as demais espécies pode contribuir na tomada de decisões quanto ao tratamento antimicrobiano mais adequado, dadas as diferenças nos limites de susceptibilidade, características clínicas e manejo terapêutico entre as cepas.(6,14)
Este trabalho teve por objetivo avaliar o perfil de sensibilidade e resistência aos antimicrobianos de Staphylococcus lugdunensis isolados de hemoculturas em um hospital de emergência no Agreste de Pernambuco, Brasil.
MATERIAL E MÉTODOS
Trata-se de um estudo laboratorial descritivo transversal, que analisou as hemoculturas obtidas do laboratório de patologia clínica de um hospital de emergência, na cidade de Caruaru, Agreste Pernambucano, no período de abril a outubro de 2015. A pesquisa foi iniciada após a aprovação pelo Comitê de Ética (Número do parecer: 1.256.386) do Centro universitário Tabosa de Almeida Asces-Unita.
As amostras de hemoculturas foram devidamente encaminhadas, em condições ideais de transporte, do laboratório de patologia clínica do hospital de emergência para o laboratório de microbiologia da Asces-Unita para os procedimentos microbiológicos necessários.
Com o auxílio de uma seringa de 3 mL, foram aspirados cerca de 0,25 mL da amostra de sangue das hemoculturas, semeados por esgotamento nos meios de cultura ágar Sangue de Carneiro e ágar MacConkey para visualização macroscópica das cepas. As placas foram incubadas em estufa a 35º-37ºC por 18 a 24 horas. Foram realizados esfregaços, posteriormente corados pelo método de Gram para confirmação microscópica.
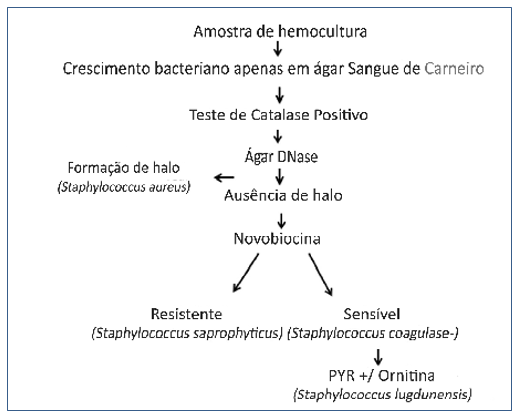
O teste de catalase foi utilizado nas amostras que apresentaram crescimento apenas em ágar Sangue de Carneiro para identificação do gênero Staphylococcus sp. Em caso de positividade, a amostra foi semeada em ágar DNase e incubada em estufa microbiológica a 35°-37°C por 18 a 24 horas para verificação da positividade da espécie Staphylococcus aureus. Caso não houvesse a formação de uma zona transparente ao redor da área de crescimento no Ágar DNase, o teste da novobiocina era realizado, onde a resistência deste determinava tratar-se de um Staphylococcus saprophyticus. No caso de sensibilidade à novobiocina, o teste de PYR (pyrrolidonil arilamidase) era realizado para diferenciação da espécie Staphylococcus lugdunensis entre as demais cepas de Staphylococcus coagulase negativa.
Para a realização do teste de PYR, o disco impregnado com L-pyrrolidonil-B-naftilamida foi colocado sobre uma lâmina e foi adicionada uma gota de água destilada estéril. Em seguida, era realizado o esfregaço com a bactéria a ser testada, sobre o disco de PYR umedecido. Após alguns minutos, era acrescentada uma gota do reativo de PYR. A reação positiva, com o desenvolvimento de cor vermelha no disco, identificava a cepa como sendo um Staphylococcus lugdunensis. (Figura 1)
As amostras, uma vez identificadas, foram submetidas ao antibiograma pela técnica de difusão em disco de Bauer e Kirby,(15) para análise do perfil de sensibilidade aos principais antibióticos de uso contra essas cepas e verificação da presença de possível resistência bacteriana. Para a realização do ensaio, inoculou-se a suspensão da bactéria em teste, ajustada ao padrão de 0,5 na escala de McFarland, sobre a superfície da placa de ágar Mueller-Hinton. Posteriormente, foram colocados discos de antimicrobianos e as placas foram incubadas em estufa bacteriológica a uma temperatura de 35-37ºC por 18 a 24 horas, sendo todo o procedimento realizado de maneira estéril.
O método foi realizado seguindo as recomendações do CLSI 2015,(16) assim como a escolha dos discos de antimicrobianos utilizados.
RESULTADOS
Do total de 95 hemoculturas analisadas, 87 (92%) mostraram crescimento bacteriano. Entre as bactérias identificadas, 60 (72%) foram classificadas como sendo do gênero Staphylococcus sp., com um quantitativo de quatro (6%) de Staphylococcus lugdunensis (Figura 2).
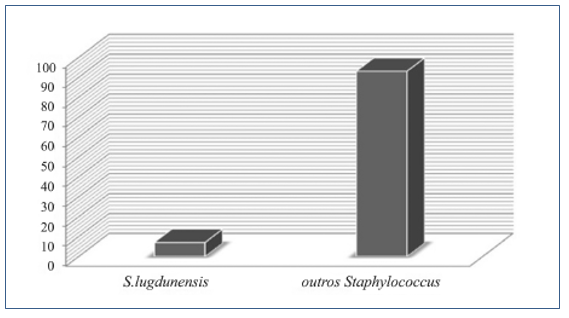
Figura 2. Prevalência do Staphylococcus lugdunensis e dos demais microrganismos Gram positivos.
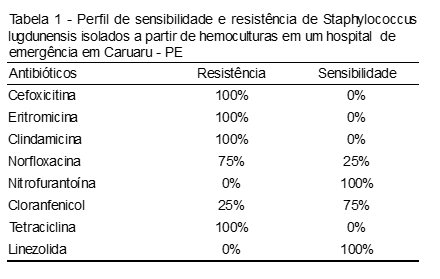
No que diz respeito ao perfil de sensibilidade e resistência aos antimicrobianos utilizados contra estes microrganismos, foi mostrado que as cepas de Staphylococcus lugdunensis apresentaram resistência significativa à cefoxitina, eritromicina, clindamicina e tetraciclina, e sensibilidade à nitrofurantoína e linezolida (Tabela 1).
DISCUSSÃO
O achado de Staphylococcus lugdunensis foi um dado importante, tendo em vista que trabalhos envolvendo esta espécie são escassos. A bactéria, apesar de detectada com menos frequência, apresenta importância clínica, com um perfil de virulência, entre os SCN, bastante próximo ao perfil dos Staphylococcus aureus, sendo considerado um patógeno emergente responsável por diversos tipos de infecções, que vão desde infecções superficiais na pele até endocardites com risco de vida.(2,7,9,12,17)
Dentre todas as amostras do gênero Staphylococcus encontradas em nosso estudo, 6% corresponderam à espécie S. lugdunensis, uma quantidade inferior à encontrada por Fadel et al.,(7) que detectaram o patógeno em 45% das amostras e, com esse achado, reforçam a necessidade dos laboratórios clínicos ficarem atentos aos isolados de SCN na perspectiva da possibilidade de isolamento de S. lugdunensis, não relacionando-o necessariamente a um organismo contaminante.
Segundo Fariña et al.,(14) em um estudo realizado com amostras clínicas de pacientes hospitalizados em ambulatório, o S. lugdunensis demonstrou uma resistência baixa a tetraciclina (0%), eritromicina (10%) e clindamicina (10%). Da mesma forma, os dados do estudo realizado por Tan et al.,(19) a partir de amostras clínicas, divergem dos dados encontrados neste estudo, demonstrando uma baixa taxa de resistência frente a tetraciclina (11,3%), eritromicina (1,8%), clindamicina (1,8%) e cefoxitina (4,7%). Estes fatos não corroboram com o presente trabalho, onde o microrganismo apresentou resistência total (100%) a estes antibióticos.
Os dados apresentados no estudo de Byrnes et al.(18) também divergem dos nossos quanto ao perfil de susceptibilidade frente a clindamicina, onde demonstrou uma maior taxa de sensibilidade (68%) em isolados hospitalares.
Os beta-lactâmicos constituem a família mais numerosa de antibióticos e a mais utilizada na antibioticoterapia. Este grupo de antimicrobianos possui em comum no seu núcleo estrutural o anel beta-lactâmico, conferindo a ele atividade bactericida, sendo usados para tratar um largo espectro de bactérias Gram-positivas e Gram-negativas.(12,13,18,20)
Alguns estudos têm demonstrado que a resistência do S. lugdunensis frente a esse grupo de antibióticos costuma ser infrequente, como relatado por Pereira et al.(13) em sua breve revisão, onde o mesmo ressalta as características microbiológicas e o perfil de susceptibilidade aos antimicrobianos frente ao S. lugdunensis, enfatizando que quase todas as cepas costumam ser sensíveis.
Porém, o aparecimento progressivo dos diversos tipos de resistência tem limitado o seu uso e a sua eficácia em determinadas situações. Em nosso estudo, a resistência aos beta-lactâmicos foi evidente, uma vez que as cepas apresentaram resistência total a 50% dos antibióticos testados. O desenvolvimento de mecanismos de resistência pode ser explicado pela produção de beta-lactamases, modificações estruturais das proteínas ligadoras de penicilina (PLP) ou diminuição da permeabilidade bacteriana ao antibiótico por mutação das porinas.(13,20)
Para entender a relevância clínica do S. lugdunensis em infecções e fornecer dados para controle e medidas epidemiológicas, é crucial a detecção e identificação confiáveis deste organismo. Vale salientar a importância da diferenciação entre os demais SCN e o S. lugdunensis, pois caso se trate deste último, o mesmo apresenta pontos de corte para interpretação do antibiograma, semelhantes aos preconizados para S. aureus e diferentes dos utilizados para os outros SCN. Sendo assim, identificações bacterianas fenotípicas errôneas de amostras clínicas podem acarretar no agravamento do quadro clínico de pacientes hospitalizados.(13,17)
CONCLUSÃO
O achado de Staphylococcus lugdunensis foi um dado relevante, uma vez que trabalhos envolvendo esta espécie são escassos. Apesar de menos frequente quando comparado aos demais do gênero Staphylococcus, o S. lugdunensis é um patógeno emergente com elevado potencial agressor.
Dessa forma, sua correta identificação laboratorial, bem como o monitoramento do perfil de sensibilidade e resistência, podem contribuir para um melhor direcionamento de medidas de controle da infecção e uso racional dos antibióticos em hospitais, minimizando assim a perda da eficácia da antibioticoterapia. Estudos como este, realizados periodicamente no ambiente hospitalar, podem auxiliar no controle das taxas de resistência, tendo em vista que dados microbiológicos orientam na escolha de uma terapêutica mais adequada para cada paciente.
Agradecimentos
Ao Laboratório de Microbiologia do Hospital Regional do Agreste, pela disponibilidade das hemoculturas analisadas, e ao Centro Universitário Tabosa de Almeida Asces-Unita, pelo apoio financeiro e disponibilidade da infraestrutura do Laboratório Escola da Instituição.
Abstract
Objective: To determine the sensibility and resistance profile of sensitivity and resistance of antimicrobials in front of Staphylococcus Lugdunensis isolated in blood cultures from an emergency hospital. Methods: Blood culture samples were aspirated and seeded in Blood sheep agar. The catalase test was performed to identify the genus Staphylococcus sp. and for the phenotypic identification of species of clinical interest, the samples were submitted to DNase, Novobiocin and PYR tests. For the analysis of the sensitivity and resistance profile, the antibiogram was used. Results: From the 95 blood cultures analyzed, 92% showed bacterial growth. Of these, 6% were identified as Staphylococcus lugdunensis, showing resistant to cefoxitin, erythromycin, clindamycin and tetracycline. Sensitivity has occurred front the nitrofurantoin and linezolid. Conclusion: The finding of Staphylococcus lugdunensis was a very important data, since works involving this species are scarce. S. lugdunensis is an emerging pathogen with high aggressive potential. Thus, its correct laboratory identification, as well as the monitoring of the susceptibility and resistance profile, can contribute with a better targeting of infection control measures, especially in hospitals.
Keywords
Staphylococcus lugdunensis; Cross infection; Antibacterial
REFERÊNCIAS
- Bellamy R, Barkham T. Staphylococcus lugdunensis infection sites: predominance of abscesses in the pelvic girdle region. Clin Infect Dis. 2002;35:32-E34.
- Sanchez AC, Flores J, Martín JM, Noria JA, Gordillo A, Hernandéz B, et al. Endocarditis infecciosa nosocomial sobre válvula nativa mitral por Staphylococcus lugdunensis. Rev Chil Cardiol. 2014;33:147-51.
- Liu PY, Huang YF, Tang CW, Chen YY, Hsieh KS, Ger LP, et al. Staphylococcus lugdunensis Infective Endocarditis: A Literature Review and Analysis of Risk Factors. J Clin Microbiol. 2010;43(6):478-84.
- Choi SH, Chung JW, Lee EJ, Kim TH, Lee MS, Kang JM, et al. Incidence, characteristics, and outcomes of Staphylococcus lugdunensis bacteremia. J Clin Microbiol. 2010;48(9):3346-9.
- Böcher S, Tonning B, Skov RL, Prag J. Staphylococcus lugdunensis, a common cause of skin and soft tissue infections in the community. J Clin Microbiol. 2009;47:946 -50.
- Shah NB, Osmon DR, Fadel H, Patel R, Kohner PC, Steckelberg JM, et al. Laboratory and Clinical Characteristics of Staphylococcus lugdunensis Prosthetic Joint Infections. J Clin Microbiol. 2010;48(5):1600-03.
- Fadel HJ, Patel R, Vetter EA, Baddour LM. Clinical Significance of a Single Staphylococcus lugdunensis-Positive Blood Culture. J Clin Microbiol. 2011;49(4):1697-99.
- Lourtet-Hascoët J, Bicart-See A, Félicé MP, Giordano G, Bonnet E. Staphylococcus lugdunensis, a serious pathogen in periprosthetic joint infections: comparison to Staphylococcus aureus and Staphylococcus epidermidis. Int J Infect Dis 2016; 51:56-61.
- Koch LD, Knoll F, Hartmann G, Lhotta K. Recurrent exit-site infection due to Staphylococcus lugdunensis – a virulent coagulase negative Staphylococcus. Perit Dial Int. 2011;31:372-3.
- Argemi X, Prévost G, Riegel P, Keller D, Meyer N, Baldeyrou M, et al. VISLISI trial, a prospective clinical study allowing identification of a new metalloprotease and putative virulence factor from Staphylococcus lugdunensis. Clin Microbiol Infect 2017; 23(5): e1-e8.
- Argemi X, Hansmann Y, Riegel P, Prévost G. Is Staphylococcus lugdunensis significant in clinical samples? J Clin Microbiol. 2017. J Clin Microbiol. 2017;55(11):3167-74.
- Silveira ACO, D’Azevedo PA. Staphylococcus lugdunensis: um olhar diferenciado no laboratório clínico. J Bras Patol Med Lab. 2011; 47:151-6.
- Pereira MFB, Carvalho RLB, Massaia IFDS, Mimica MJ. Staphylococcus lugdunensis: um patógeno emergente em humanos. Arq Med Hosp Fac Cienc Med. 2013;58(3):151.
- Fariña N, Carpinelli L, Samudio M, Guillén R, Laspina F, Sanabria R, et al. Staphylococcus coagulasa-negativa clínicamente significativos. Especies más frecuentes y factores de virulência. Rev Chil Infectol. 2013;30(5):480-8.
- Piaskowski CA, Yamanaka EHU, Romanel M. Manual para antibiograma. Técnica de difusão por disco. Laborclin produtos para laboratórios Ltda. Pinhais – PR. Maio, 2007.
- Clinical and Laboratory Standards Institute. Performance Standards for Antimicrobial Susceptibility Testing. Twenty-Second Informational Supplement M100-S22, 2015;35(3).
- Pereira EM, Oliveira FL, Schuenck RP, Zoletti GO, dos Santos KR. Detection of Staphylococcus lugdunensis by a new species-specific PCR based on the fbl gene. FEMS Immunol Med Microbiol. 2010;58(2):295-8.
- Byrnes TJ, Rose BT, Myers NM, Myers J. Staphylococcus lugdunensis Bacteremia in Adults in a Large Community Teaching Hospital. Report of 29 Episodes and Review of its Epidemiology, Microbiology, Clinical Manifestations, and Treatment. J Medical Microbiol Diagnosis. 2014;S2.
- Tan TY, Ng SY, He J. Microbiological characteristics, presumptive identification, and antibiotic susceptibilities of Staphylococcus lugdunensis. J Clin Microbiol. 2008;46(7):2393-5.
- Cooke RP, James SE, Sallomi DF. Infective discitis due to Staphylococcus lugdunensis-a case of missed opportunity. Br J Biomed Sci 2003; 60(3):162-4.
Correspondência
Danielle Guimarães Araújo
Avenida Portugal nº 584, Bairro Universitário
55016-400 – Caruaru-PE, Brasil
Fone: (81) 2103-2000
